Bei Patienten mit entzündlicher Darmerkrankung können Antibiotika ein zweischneidiges Schwert sein. Die häufig für Darmflackern verschriebenen Breitbandmedikamente können hilfreiche Mikroben zusammen mit schädlichen abtöten, was sich manchmal im Laufe der Zeit verschlechtern. Beim Kampf gegen Darmentzündung möchten Sie nicht immer einen Vorschlaghammer zu einem Messerkampf bringen.
Forscher des MIT -Labors für Informatik und künstliche Intelligenz (CSAIL) und die McMaster College haben eine neue Verbindung identifiziert Das erfordert einen gezielteren Ansatz. Das Molekül, Enterololin genannt, unterdrückt eine Gruppe von Bakterien, die mit Morbus Crohn-Aufflackern verbunden sind, während der Relaxation des Mikrobioms weitgehend intakt bleibt. Mit einem generativen KI -Modell kartierte das Staff, wie die Verbindung funktioniert, ein Prozess, der normalerweise Jahre dauert, aber hier auf nur wenige Monate beschleunigt wurde.
„Diese Entdeckung spricht für eine zentrale Herausforderung in der Antibiotika -Entwicklung“, sagt Jon Stokes, Senior Autor von A. Neues Papier über die ArbeitAssistenzprofessor für Biochemie und Biomedizinische Wissenschaften bei McMaster und Forschungsunternehmen in der Abdul Latif Jameel -Klinik der MIT für maschinelles Lernen im Gesundheitswesen. „Das Drawback findet keine Moleküle, die Bakterien in einer Schüssel töten-wir haben das schon lange tun. Eine große Hürde besteht darin, herauszufinden, was diese Moleküle tatsächlich innerhalb von Bakterien tun. Ohne dieses detaillierte Verständnis können Sie diese Frühstadien-Antibiotika nicht in sichere und wirksame Therapien für Patienten entwickeln.“
Enterololin ist ein Schritt in Richtung Präzisionsantibiotika: Behandlungen, die nur die Bakterien ausschalten, die Probleme verursachen. In Mausmodellen der Mausentzündung wurde das Arzneimittel auf ein ON aufgenommen Escherichia coliein Darmbakterium, das die Fackeln verschlimmern kann, während die meisten anderen mikrobiellen Bewohner unberührt werden. Mäuse erholten sich schneller und behielten ein gesünderes Mikrobiom bei als diejenigen, die mit Vancomycin behandelt wurden, einem gemeinsamen Antibiotikum.
Das molekulare Ziel, das es in Bakterienzellen bindet, erfordert normalerweise jahrelange mühsame Experimente, die den Wirkungsmechanismus eines Arzneimittels abbiegt. Das Labor von Stokes entdeckte Enterololin mit einem Excessive-Throughput-Screening-Ansatz, aber das Bestimmen seines Ziels wäre der Engpass gewesen. Hier wandte sich das Staff zu Diffdockein generatives KI -Modell, das von MIT PhD -Scholar Gabriele Corso und MIT -Professor Regina Barzilay entwickelt wurde.
Diffdock wurde entwickelt, um vorherzusagen, wie kleine Moleküle in die Bindungstaschen von Proteinen passen, ein notorisch schwieriges Drawback in der Strukturbiologie. Traditionelle Docking -Algorithmen suchen nach möglichen Orientierungen unter Verwendung von Bewertungsregeln und erzielen häufig laute Ergebnisse. Diffdock Stattdessen dockendocken als probabilistisches Argumentationsproblem: Ein Diffusionsmodell verfeinert iterativ Vermutungen, bis es im wahrscheinlichsten Bindungsmodus konvergiert.
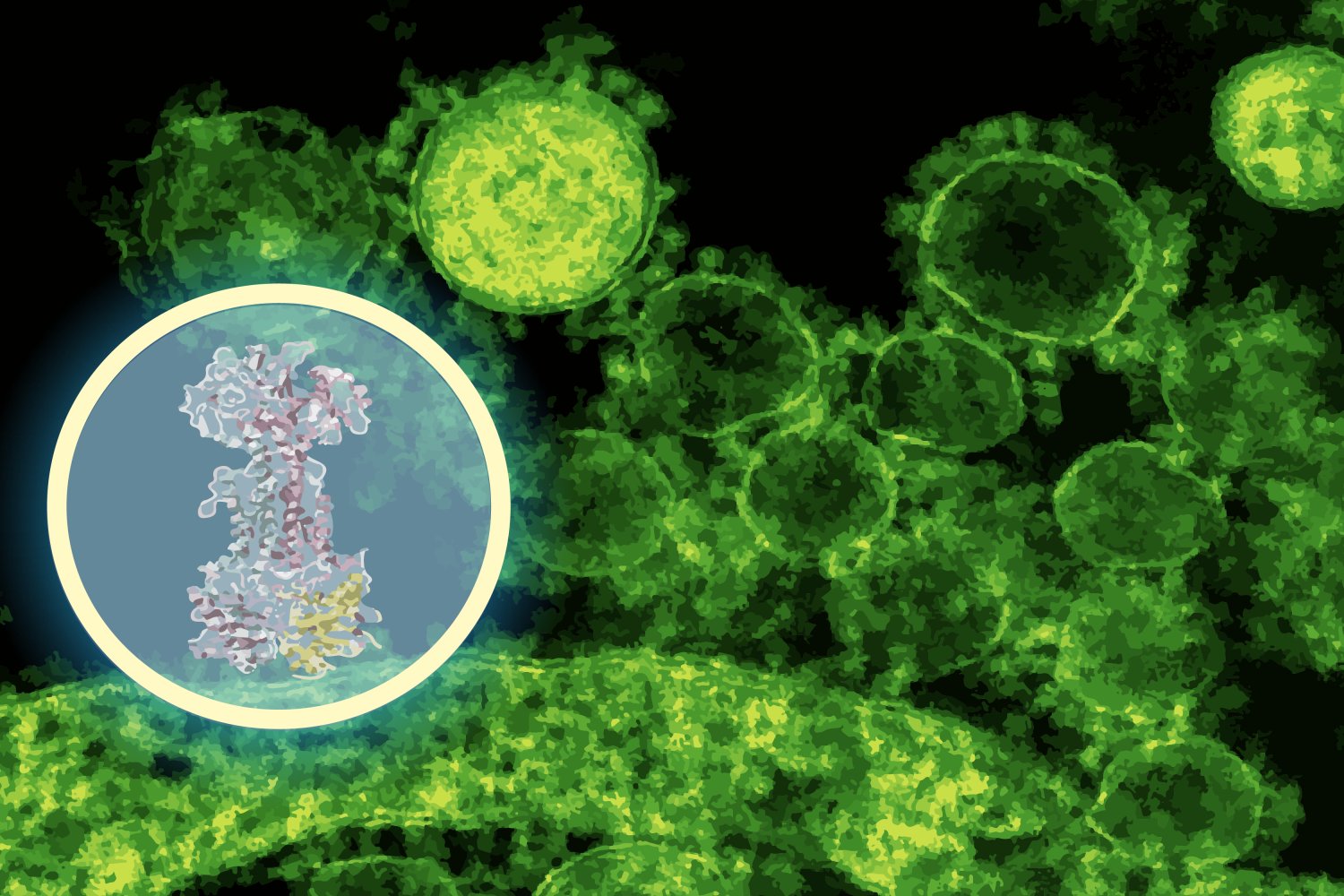
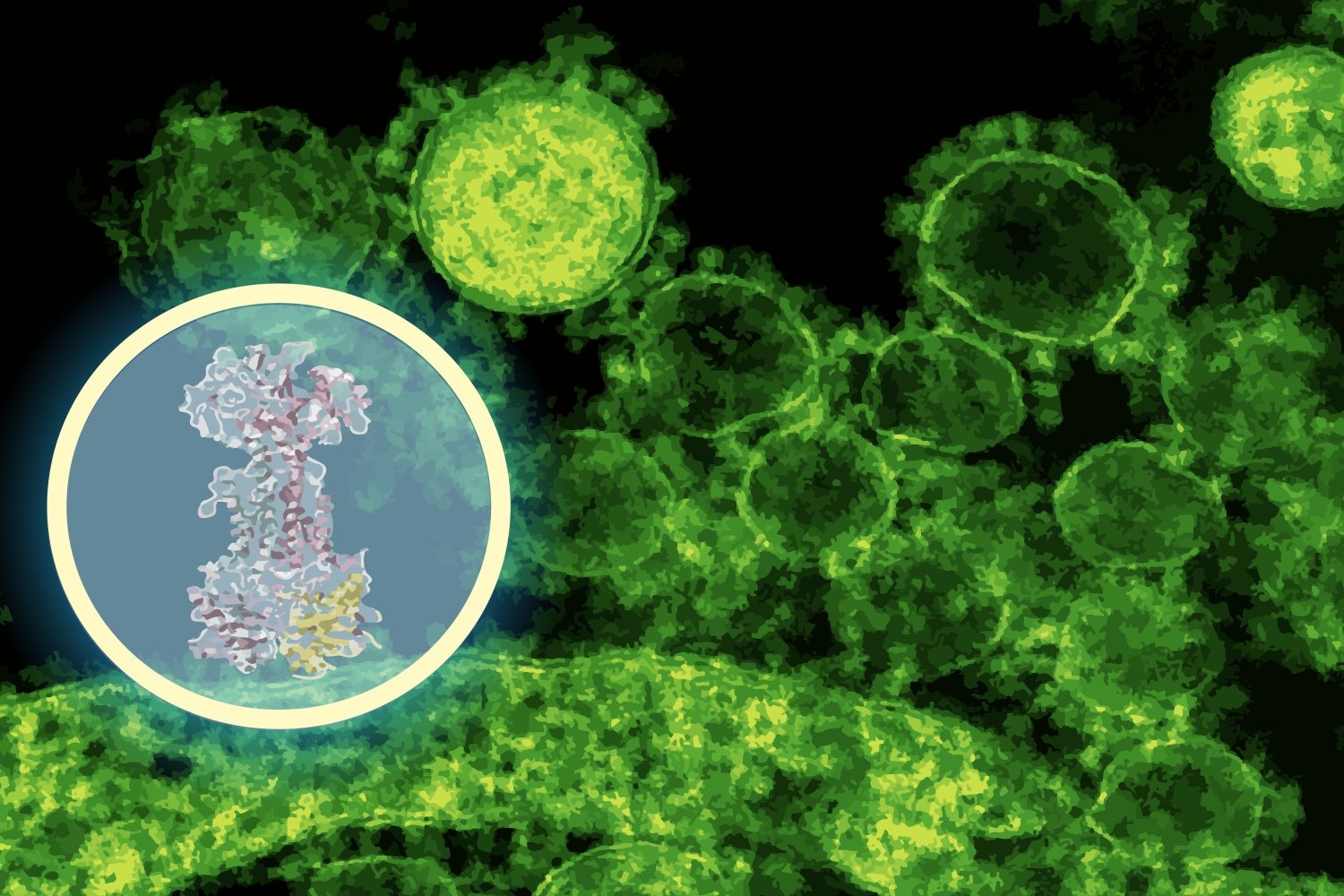
„In nur wenigen Minuten sagte das Modell voraus, dass Enterololin an einen Proteinkomplex namens LOLCDE bindet, was für den Transport von Lipoproteinen in bestimmten Bakterien essentiell ist“, sagt Barzilay, der auch die Jameel-Klinik mit führt. „Das warfare ein sehr konkreter Vorsprung – einer, der Experimente leiten konnte, anstatt sie zu ersetzen.“
Stokes ‚Gruppe hat diese Vorhersage dann auf die Prüfung gebracht. Unter Verwendung von Diffdock-Vorhersagen als experimentelles GPS entwickelten sie zuerst Enterololin-resistente Mutanten von E. coli im Labor, das sich ergab, dass Veränderungen in der DNA der Mutante, die Lolcde zugeordnet warfare, genau dort, wo Diffdock vorhergesagt hatte, das Enterololin zu binden hatte. Sie führten auch eine RNA -Sequenzierung durch, um festzustellen, welche bakteriellen Gene beim Medikament ausgesetzt oder ausgeschaltet wurden, und verwendeten CRISPR, um die Expression des erwarteten Ziels selektiv niederzuschlagen. Diese Laborexperimente zeigten alle Störungen in Wegen, die mit dem Lipoproteintransport gebunden waren, genau das, was Diffdock vorhergesagt hatte.
„Wenn Sie das Computermodell und die nasslabenden Daten sehen, die auf denselben Mechanismus hinweisen, glauben Sie, dass Sie etwas herausgefunden haben“, sagt Stokes.
Für Barzilay hebt das Projekt eine Verschiebung der Verwendung von KI in den Biowissenschaften hervor. „Bei einer Menge KI -Verwendung bei der Entdeckung von Arzneimitteln ging es darum, den chemischen Raum zu durchsuchen und neue Moleküle zu identifizieren, die aktiv sein könnten“, sagt sie. „Wir zeigen hier, dass KI auch mechanistische Erklärungen liefern kann, die für die Verschiebung eines Moleküls durch die Entwicklungspipeline von entscheidender Bedeutung sind.“
Diese Unterscheidung ist wichtig, weil Studien der Aktionsverkleidung häufig ein wesentlicher Fee-limitierender Schritt in der Arzneimittelentwicklung sind. Traditionelle Ansätze können 18 Monate bis zwei Jahre oder mehr dauern und Millionen von Greenback kosten. In diesem Fall verkürzte das MIT -MCMaster -Staff den Zeitplan auf etwa sechs Monate zu einem Bruchteil der Kosten.
Enterololin befindet sich noch in den frühen Entwicklungsstadien, die Übersetzung ist jedoch bereits im Gange. Stokes ‚Spinout Firm, Stoked Bio, hat die Verbindung lizenziert und optimiert ihre Eigenschaften für potenzielle menschliche Verwendung. Frühe Arbeiten untersuchen auch Derivate des Moleküls gegen andere resistente Krankheitserreger, wie z. Klebsiella pneumoniae. Wenn alles intestine geht, könnten klinische Studien in den nächsten Jahren beginnen.
Die Forscher sehen auch breitere Auswirkungen. Antibiotika mit schmalem Spektrum sind seit langem als eine Möglichkeit, Infektionen ohne Kollateralschäden am Mikrobiom zu behandeln, aber es warfare schwer zu entdecken und zu validieren. KI -Instruments wie Diffdock könnten diesen Prozess praktischer machen und schnell eine neue Era gezielter antimikrobieller Era ermöglichen.
Bei Patienten mit Murren und anderen entzündlichen Darmerkrankungen kann die Aussicht auf ein Medikament, das die Symptome reduziert, ohne das Mikrobiom zu destabilisieren, eine sinnvolle Verbesserung der Lebensqualität bedeuten. Im größeren Bild können Präzisionsantibiotika dazu beitragen, die wachsende Bedrohung durch antimikrobielle Resistenz zu bekämpfen.
„Was mich erregt, ist nicht nur diese Verbindung, sondern auch die Idee, über den Mechanismus der Wirkungsaufklärung nachzudenken, als etwas, das wir schneller tun können, mit der richtigen Kombination aus KI, menschlicher Instinct und Laborversuche“, sagt Stokes. „Das hat das Potenzial, die Entdeckung der Drogen für viele Krankheiten zu verändern, nicht nur von Crohn.“
„Eine der größten Herausforderungen für unsere Gesundheit ist die Zunahme von antimikrobiell-resistenten Bakterien, die selbst unseren besten Antibiotika ausweichen“, fügt Yves Brun, Professor an der Universität von Montreal, und an der Distinguished Professor an der Indiana College Bloomington hinzu, die nicht in die Zeitung involviert warfare. „KI wird zu einem wichtigen Instrument in unserem Kampf gegen diese Bakterien. Diese Studie verwendet eine leistungsstarke und elegante Kombination von AI -Methoden, um den Wirkungsmechanismus eines neuen Antibiotika -Kandidaten zu bestimmen, ein wichtiger Schritt in seiner potenziellen Entwicklung als therapeutisch.“
Corso, Barzilay und Stokes schrieben die Zeitung mit den McMaster -Forschern Denise B. Catacutan, Vian Tran, Jeremie Alexander, Yeganeh Yousefi, Megan Tu, Stewart McLellan und Dominique Tertigas sowie Professoren Jakob Magolan, Michael Surette, Eric Brown und Brian Coombes. Ihre Forschung wurde teilweise von der Weston Household Basis unterstützt. das David Braley Middle für Antibiotika -Entdeckung; die kanadischen Institute für Gesundheitsforschung; der Pure Sciences and Engineering Analysis Council von Kanada; M. und M. Heersink; Kanadische Institute für Gesundheitsforschung; Ontario Graduate Stipendienpreis; die Jameel -Klinik; und die Entdeckung der medizinischen Gegenmaßnahmen gegen das neue und aufkommende Bedrohungsprogramm der US Defence Menace Discount Company.
Die Forscher haben Sequenzierungsdaten in öffentlichen Repositories veröffentlicht und den Diffdock-L-Code offen auf GitHub veröffentlicht.