Eine enorme Suche nach natürlicher Vielfalt hat Wissenschaftler des MCGovern -Instituts für Gehirnforschung des MIT und des Broad Institute of MIT und Harvard dazu veranlasst, alte Systeme aufzudecken, um das Genom -Bearbeitungs -Toolbox zu erweitern.
Diese Systeme, die die Forscher TIGR (Tandem Interspaced Information RNA) -Systeme bezeichnen, verwenden sie RNA, um sie an bestimmten Stellen auf DNA zu führen. TIGR -Systeme können neu programmiert werden, um auf eine DNA -Sequenz von Interesse abzuzielen, und sie haben unterschiedliche funktionelle Module, die auf die gezielte DNA wirken können. Zusätzlich zu seiner Modularität ist TIGR im Vergleich zu anderen RNA-geführten Systemen wie CRISPR sehr kompakt, was ein großer Vorteil für die Bereitstellung von therapeutischem Kontext ist.
Diese Ergebnisse sind On-line gemeldet am 27. Februar in der Zeitschrift Wissenschaft.
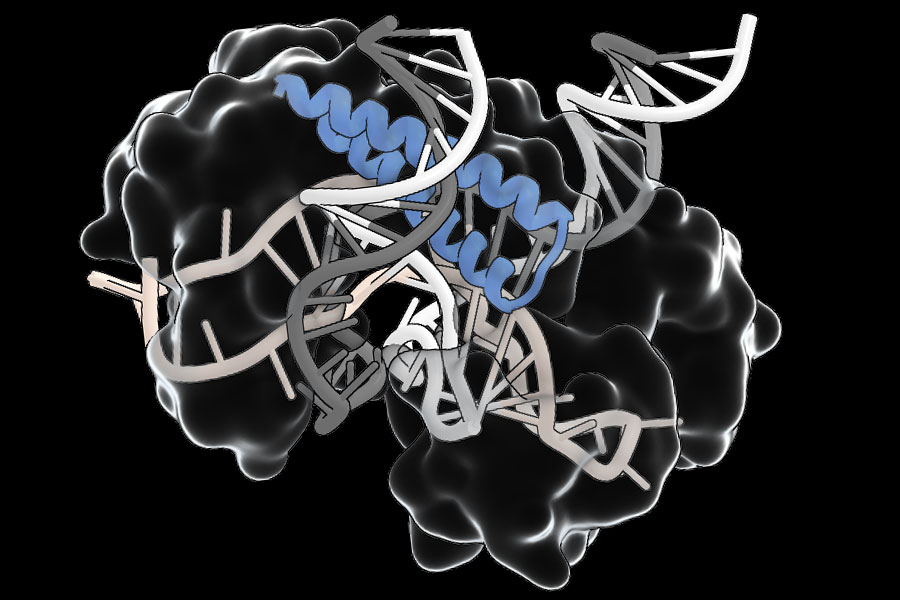
„Dies ist ein sehr vielseitiges RNA-gesteuerter System mit vielen unterschiedlichen Funktionen“, sagt Feng Zhang, Professor für Neurowissenschaften von James und Patricia Poitras am MIT, der die Forschung leitete. Die TIGR-assoziierten (TAS) Proteine, die Zhangs Workforce fand, teilen eine charakteristische RNA-bindende Komponente, die mit einer RNA-Anleitung interagiert, die sie an eine bestimmte Stelle im Genom leitet. Einige schneiden die DNA an dieser Stelle unter Verwendung eines benachbarten DNA-Schnittsegments des Proteins. Diese Modularität könnte die Werkzeugentwicklung erleichtern und es Forschern ermöglichen, nützliche neue Merkmale in natürliche TAS -Proteine auszutauschen.
„Die Natur ist ziemlich unglaublich“, sagt Zhang, der auch Ermittler am McGovern-Institut und das Howard Hughes Medical Institute ist, ein Kernmitglied des Broad Institute, Professor für Gehirn- und Kognitive Wissenschaften und Organic Engineering bei MIT und Co-Direktor der Okay. lisa yang und Hock E. Tan E. Tan Middle for Molekulartherapie bei MIT. „Es hat eine enorme Menge an Vielfalt, und wir haben diese natürliche Vielfalt untersucht, um neue biologische Mechanismen zu finden und sie für verschiedene Anwendungen zur Manipulation biologischer Prozesse zu nutzen“, sagt er. Zuvor adaptierte Zhangs Workforce bakterielle CRISPR -Systeme in Gen -Bearbeitungswerkzeuge, die die moderne Biologie verändert haben. Sein Workforce hat auch eine Vielzahl programmierbarer Proteine gefunden, sowohl von CRISPR -Systemen als auch darüber hinaus.
In ihren neuen Arbeiten, um neuartige programmierbare Systeme zu finden, begann das Workforce zunächst in einem strukturellen Merkmal des CRISPR-Cas9-Proteins, das an den RNA-Leitfaden des Enzyms bindet. Dies ist ein Schlüsselmerkmal, das Cas9 zu einem so leistungsstarken Werkzeug gemacht hat: „RNA-geführtes macht es relativ einfach, neu zu programmieren, da wir wissen, wie RNA an andere DNA oder andere RNA bindet“, erklärt Zhang. Sein Workforce suchte Hunderte Millionen biologischer Proteine mit bekannten oder vorhergesagten Strukturen und suchte nach einem ähnlichen Bereich. Um weiter verwandte Proteine zu finden, verwendeten sie einen iterativen Prozess: Aus Cas9 identifizierten sie ein Protein namens IS110, das zuvor von anderen zur Bindung von RNA gezeigt worden conflict. Anschließend haben sie die strukturellen Merkmale von IS110 aufgenommen, die die RNA -Bindung ermöglichen und ihre Suche wiederholten.
Zu diesem Zeitpunkt hatte die Suche so viele entfernt verwandte Proteine aufgetaucht, dass sie sich der künstlichen Intelligenz zugewandt haben, um die Liste zu verstehen. „Wenn Sie iterative, tiefe Bergbildung durchführen, können die daraus resultierenden Treffer so unterschiedlich sein, dass sie mithilfe von standardmäßigen phylogenetischen Methoden schwer analysieren können, die auf konservierter Sequenz beruhen“, erklärt Guilhem Faure, ein Computerbiologe in Zhangs Labor. Mit einem Protein -Großsprachmodell conflict das Workforce in der Lage, die Proteine zu gruppieren, die sie nach ihren wahrscheinlichen evolutionären Beziehungen in Gruppen gefunden hatten. Eine Gruppe unterscheidet sich von den anderen, und ihre Mitglieder waren besonders faszinierend, da sie von Genen mit regelmäßig verteilten sich wiederholenden Sequenzen kodiert wurden, die an eine wesentliche Komponente von CRISPR -Systemen erinnern. Dies waren die Tig-Tas-Systeme.
Das Workforce von Zhang entdeckte mehr als 20.000 verschiedene TAS-Proteine, die hauptsächlich in Bakterien-infizierenden Viren auftraten. Sequenzen innerhalb der sich wiederholenden Area jedes Gens-ihre TIGR-Arrays-codieren eine RNA-Anleitung, die mit dem RNA-bindenden Teil des Proteins interagiert. In einigen Fällen befindet sich die RNA-Bindungsregion neben einem DNA-Schneidenteil des Proteins. Andere scheinen an andere Proteine zu binden, was darauf hindeutet, dass sie diese Proteine auf DNA -Ziele richten könnten.
Zhang und sein Workforce experimentierten mit Dutzenden von TAS -Proteinen und zeigten, dass einige programmiert werden können, um zielgerichtete Schnitte in menschlichen Zellen vorzunehmen. Wenn sie darüber nachdenken, Tig-Tas-Systeme in programmierbare Instruments zu entwickeln, werden die Forscher von Funktionen ermutigt, die diese Instruments besonders flexibel und präzise machen könnten.
Sie stellen fest, dass CRISPR -Systeme nur auf DNA -Segmente gerichtet werden können, die von kurzen Motiven flankiert werden, die als PAMs (Protospacer benachbarte Motive) bekannt sind. Tig Tas -Proteine dagegen haben keine solche Anforderung. „Dies bedeutet theoretisch, dass jeder Standort im Genom zielgerichtet sein sollte“, sagt der wissenschaftliche Berater Rhiannon Macrae. Die Experimente des Groups zeigen auch, dass Tig-Systeme eine „Twin-Information-System“ bezeichnen und mit beiden Strängen der DNA-Doppelhelix interagieren, um in ihren Zielsequenzen nach Hause zu gehen, was sicherstellen sollte, dass sie nur dort handeln, wo sie von ihrem RNA-Leitfaden geleitet werden. Darüber hinaus sind TAS -Proteine kompakt – ein Viertel der Größe CAS9 im Durchschnitt -, was sie leichter liefern lässt, was ein wichtiges Hindernis für den therapeutischen Einsatz von Genbearbeitungswerkzeugen überwinden könnte.
Das Workforce von Zhang von ihrer Entdeckung untersucht nun die natürliche Rolle von Tigsystemen in Viren sowie die Artwork und Weise, wie sie für Forschung oder Therapeutika angepasst werden können. Sie haben die molekulare Struktur eines der TAS -Proteine bestimmt, die sie in menschlichen Zellen funktionierten, und werden diese Informationen verwenden, um ihre Bemühungen zu leiten, um sie effizienter zu gestalten. Darüber hinaus stellen sie Verbindungen zwischen Tig-Tas-Systemen und bestimmten RNA-Verarbeitungsproteinen in menschlichen Zellen fest. „Ich denke, es gibt noch mehr, um zu studieren, was einige dieser Beziehungen sein mögen, und es kann uns helfen, besser zu verstehen, wie diese Systeme beim Menschen verwendet werden“, sagt Zhang.
Dieses Werk wurde von der Helen Hay Whitney Basis, dem Howard Hughes Medical Institute, dem Okay. Lisa Yang und dem Hock E. Tan Middle for Molekular Therapeutics, dem breiten Institut -Therapeutika -Geschenkspender, der Pershing Sq. Basis, der William Ackman, der Neri Oxman, der Phillips Household, J. und P. Poitras sowie der BT Charable Basis.