()
Der Artikel wurde von Guanao Yan, Ph.D. Pupil der Statistik und Datenwissenschaft an der UCLA. Guanao ist der erste Autor des Artikels von Nature Communications Evaluate Artikel (1).
Räumlich aufgelöste Transkriptomik (SRT) revolutionieren Genomik Durch Aktivieren der Hochdurchsatzmessung der Genexpression und bei der Erhaltung des räumlichen Kontextes. Im Gegensatz zur Einzelzell-RNA-Sequenzierung (SCRNA-seq), die Transkriptome ohne räumliche Ortsinformationen erfasst, ermöglicht SRT Forschern, die Genexpression genau zu kartieren, um genaue Stellen innerhalb eines Gewebes zu erhalten, wodurch Einblicke in die Gewebeorganisation, die zellulären Wechselwirkungen und die räumlich koordinierte Genaktivität geliefert werden. Das zunehmende Volumen und die Komplexität von SRT -Daten erfordern die Entwicklung robuster statistischer und rechnerischer Methoden, wodurch dieses Feld für Datenwissenschaftler, Statistiker und maschinelle Lernen (ML) von großer Bedeutung ist. Techniken wie räumliche Statistiken, grafische Modelle und Deep-Lernen wurden angewendet, um aus diesen Daten aussagekräftige biologische Erkenntnisse zu extrahieren.
Ein wichtiger Schritt in der SRT-Analyse ist der Nachweis räumlich variabler Gene (SVGs)-Genen, deren Expression nicht randomisch über räumliche Stellen variiert. Die Identifizierung von SVGs ist entscheidend für die Charakterisierung der Gewebearchitektur, der funktionellen Genmodule und der zellulären Heterogenität. Trotz der raschen Entwicklung von Computermethoden für die SVG -Erkennung variieren diese Methoden in ihren Definitionen und statistischen Rahmenbedingungen stark, was zu inkonsistenten Ergebnissen und Herausforderungen bei der Interpretation führt.
In unserer kürzlich veröffentlichten Rezension veröffentlicht in Naturkommunikation (1) haben wir systematisch 34 von Experten begutachtete SVG-Erkennungsmethoden untersucht und ein Klassifizierungsrahmen eingeführt, das die biologische Bedeutung verschiedener SVG-Typen verdeutlicht. Dieser Artikel bietet einen Überblick über unsere Ergebnisse und konzentriert sich auf die drei Hauptkategorien von SVGs und die statistischen Prinzipien, die ihrer Erkennung zugrunde liegen.

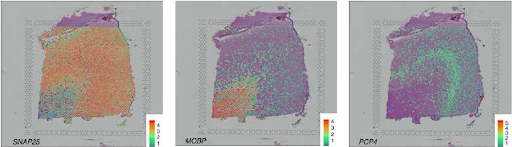
SVG -Erkennungsmethoden zielen darauf ab, Gene aufzudecken, deren räumliche Expression eher biologische Muster als technische Rauschen widerspiegelt. Basierend auf unserer Überprüfung von 34 von Experten begutachteten Methoden kategorisieren wir SVGs in drei Gruppen: Gesamt-SVGs, zelltypspezifische SVGs und SVGs mit räumlicher Domänen-Marker (Abbildung 2).

Methoden zum Nachweis der drei SVG -Kategorien dienen unterschiedlichen Zwecken (Abb. 3). Erstens der Nachweis von Gesamtscreenings informativen Genen für nachgeschaltete Analysen, einschließlich der Identifizierung räumlicher Domänen und funktioneller Genmodule. Zweitens zielt das Nachweis von SVGs vom Zelltyp darauf ab, räumliche Variationen innerhalb eines Zelltyps aufzudecken und verschiedene Zellsubpopulationen oder Zustände innerhalb der Zelltypen zu identifizieren. Drittens wird der SVG-Erkennung von räumlicher Domänen-Marker verwendet, um Marker-Gene zu finden, um die bereits nachgewiesenen räumlichen Domänen zu kommentieren und zu interpretieren. Diese Marker helfen dabei, die molekularen Mechanismen zu verstehen, die räumlichen Domänen zugrunde liegen, und helfen bei der Annotation von Gewebeschichten in anderen Datensätzen.

Die Beziehung zwischen den drei SVG -Kategorien hängt von den Erkennungsmethoden ab, insbesondere den Null- und alternativen Hypothesen, die sie einsetzen. Wenn eine Gesamt-SVG-Erkennungsmethode die Nullhypothese verwendet, dass die Expression eines Nicht-SVG unabhängig von der räumlichen Lage und der alternativen Hypothese ist, dass jede Abweichung von dieser Unabhängigkeit auf ein SVG angibt, sollten seine SVGs theoretisch sowohl zelltypspezifische SVGs als auch räumlich einschließen -Area-Marker SVGs. Beispielsweise ist Despace (2) eine Methode, die sowohl Gesamt-SVGs als auch räumliche Domänen-Marker-SVGs erkennt, und seine nachgewiesenen Gesamt-SVGs müssen Marker-Gene für einige räumliche Domänen sein. Diese Einschlussbeziehung gilt außer in extremen Szenarien, wie z. Wenn jedoch die various Hypothese einer allgemeinen SVG-Erkennungsmethode für ein spezifisches räumliches Expressionsmuster definiert ist, enthalten seine SVGs möglicherweise keine zellspezifischen SVGs oder räumlichen Domänen-Marker-SVGs.
Um zu verstehen, wie SVGs erkannt werden, haben wir die statistischen Ansätze in drei Haupttypen von Hypothesentests eingeteilt:
- Abhängigkeitstest – Untersucht die Abhängigkeit zwischen dem Expressionsniveau eines Gens und dem räumlichen Ort.
- Regression Fixe-Impact-Take a look at-Untersucht, ob einige oder alle mit festen Effekten Kovariaten, beispielsweise der räumliche Ort, zum Mittelwert der Antwortvariablen beitragen, dh die Expression eines Gens.
- Regression Zufallseffekt-Take a look at (Varianzkomponententest)-Untersucht, ob der Zufallseffekt, beispielsweise der räumliche Ort, zur Varianz der Antwortvariablen, dh der Expression eines Gens, beiträgt.
Um weiter zu erklären, wie diese Assessments für den SVG -Nachweis verwendet werden, bezeichnen wir 𝑌 als Expressionsniveau des Gens und 𝑆 als räumliche Stellen. Der Abhängigkeitstest ist der allgemeinste Hypothesentest für die SVG -Erkennung. Für ein bestimmtes Gen entscheidet es, ob das Expressionsniveau des Gens 𝑌 unabhängig vom räumlichen Ort 𝑆 ist, dh die Nullhypothese lautet:

Es gibt zwei Arten von Regressionstests: Assessments mit festen Effekten, bei denen angenommen wird, dass die Auswirkung des räumlichen Ortes fest ist, und Zufallseffekttests, die die Wirkung des räumlichen Ortes als zufällig annehmen. Um diese beiden Testtypen zu erklären, verwenden wir ein lineares gemischtes Modell für ein bestimmtes Gen als Beispiel:

wobei die Antwortvariable (y_i ) das Expressionsniveau des Gens an Spot (i ) ist, (x_i ) ( epsilon ) (r^p ) zeigt die festen Effekt-Kovariaten von Spot ((R^p ) an i ), (z_i ) ( epsilon ) (r^q ) bezeichnet die zufällige Effekt-Kovariaten von Spot (i ) und ( epsilon_i ) ist der zufällige Messfehler am Spot (i ) mit null Mittelwert. In den Modellparametern ist ( beta_0 ) der (fixe) Intercept, ( Beta ) ( epsilon ) (r^p ) Zeigt die festen Effekte an und ( gamma ) ( epsilon ) (r^q ) bezeichnet die zufälligen Effekte mit Nullmitteln und der Kovarianzmatrix:

In diesem linearen gemischten Modell wird die Unabhängigkeit zwischen Zufallseffekt und zufälligen Fehlern und zwischen zufälligen Fehlern angenommen.
Assessments mit festen Wirkung untersuchen S) Tragen Sie zum Mittelwert der Antwortvariablen bei. Wenn alle Kovariaten mit festen Effekten keinen Beitrag leisten, dann:

Die Nullhypothese

impliziert

Zufällige Wirkungstests untersuchen, ob die Zufallseffekt-Kovariaten (z_i ) (abhängig von räumlichen Stellen abhängen S) Tragen Sie zur Varianz der Antwortvariablen varyi bei und konzentrieren sich auf die Zersetzung:

und Assessments, ob der Beitrag der Zufallseffekt-Kovariaten Null ist. Die Nullhypothese:

impliziert

Unter den 23 Methoden, die Frequentist-Hypothesentests verwenden, wurden hauptsächlich Abhängigkeitstests und Zufallseffekt-Regressionstests angewendet, um Gesamt-SVGs nachzuweisen, während in allen drei SVG-Kategorien eine Regressionstests mit festen Effekten verwendet wurden. Das Verständnis dieser Unterscheidungen ist der Schlüssel zur Auswahl der richtigen Methode für bestimmte Forschungsfragen.
Durch die Verbesserung der SVG -Erkennungsmethoden müssen die Erkennungsleistung, die Spezifität und die Skalierbarkeit ausbalancieren und gleichzeitig wichtige Herausforderungen bei der räumlichen Transkriptomikanalyse angegangen werden. Zukünftige Entwicklungen sollten sich auf die Anpassung von Methoden an verschiedene SRT-Technologien und Gewebetypen konzentrieren und die Unterstützung für SRT-Daten mehrerer Proben erweitern, um die biologischen Erkenntnisse zu verbessern. Darüber hinaus ist die Stärkung der statistischen Strenge und Validierungsrahmen für die Gewährleistung der Zuverlässigkeit der SVG -Erkennung von entscheidender Bedeutung. Benchmarking -Studien müssen ebenfalls verfeinert werden, mit klareren Bewertungsmetriken und standardisierten Datensätzen, um robuste Methodenvergleiche bereitzustellen.
Referenzen
(1) Yan, G., Hua, SH & Li, JJ (2025). Kategorisierung von 34 Rechenmethoden zum Nachweis räumlich variabler Gene aus räumlich aufgelösten Transkriptomik -Daten. Naturkommunikation16, 1141. https://doi.org/10.1038/s41467-025-56080-w
(2) Cai, P., Robinson, MD & Tiberi, S. (2024). Despace: Räumlich variabler Generkennung durch unterschiedliche Expressionstests räumlicher Cluster. Bioinformatik, 40 (2). https://doi.org/10.1093/bioinformatics/btae027