Wissenschaftler des McGovern-Instituts für Gehirnforschung am MIT und des Broad Institute of MIT und Harvard haben ein kompaktes RNA-gesteuerter Enzym überarbeitet, das sie in Bakterien zu einem effizienten, programmierbaren Herausgeber der menschlichen DNA gefunden haben.
Das von ihnen erstellte Protein, das NovaISCB genannt wurde, kann angepasst werden, um genaue Änderungen am genetischen Code vorzunehmen, die Aktivität spezifischer Gene zu modulieren oder andere Bearbeitungsaufgaben auszuführen. Da seine geringe Größe die Lieferung an Zellen vereinfacht, sagen die Entwickler von Novaiscb, dass es ein vielversprechender Kandidat für die Entwicklung von Gentherapien zur Behandlung oder Vorbeugung von Krankheiten ist.
Die Studie wurde von durchgeführt Feng ZhangDer Professor für Neurowissenschaften von James und Patricia Poitras am MIT, der auch Forscher am McGovern -Institut und Howard Hughes Medical Institute und Kernmitglied des Broad Institute ist. Zhang und sein Workforce meldeten diesen Monat ihre Open-Entry-Arbeit in der Zeitschrift Naturbiotechnologie.
Novaiscb wird von einem bakteriellen DNA-Cutter abgeleitet, der zu einer Familie von Proteinen gehört, die ISCBS namens ISCBS, die Zhangs Labor 2021 entdeckt hat. Wie Cas9 schneiden ISCB -Enzyme die DNA an den von einer RNA -Führung angegebenen Stellen. Durch die Neuprogrammierung dieses Leitfadens können Forscher die Enzyme auf Sequenzen ihrer Wahl umleiten.
ISCBS hatte die Aufmerksamkeit des Groups nicht nur, weil es wichtige Merkmale von CRISPRs DNA-Schneidungs-Cas9 teilt, sondern auch, weil sie ein Drittel seiner Größe sind. Dies wäre ein Vorteil für potenzielle Gentherapien: Kompakte Instruments sind leichter zu Zellen zu liefern, und mit einem kleinen Enzym hätten Forscher mehr Flexibilität für Bastler, was möglicherweise neue Funktionen hinzufügt, ohne Instruments zu erstellen, die für den klinischen Einsatz zu sperrig waren.
Aus ihren ersten Studien an ISCBs wussten Forscher in Zhangs Labor, dass einige Familienmitglieder DNA -Ziele in menschlichen Zellen senken konnten. Keiner der bakteriellen Proteine funktionierte jedoch intestine genug, um therapeutisch eingesetzt zu werden: Das Workforce müsste ein ISCB ändern, um sicherzustellen, dass die Ziele in menschlichen Zellen effizient bearbeiten können, ohne den Relaxation des Genoms zu stören.
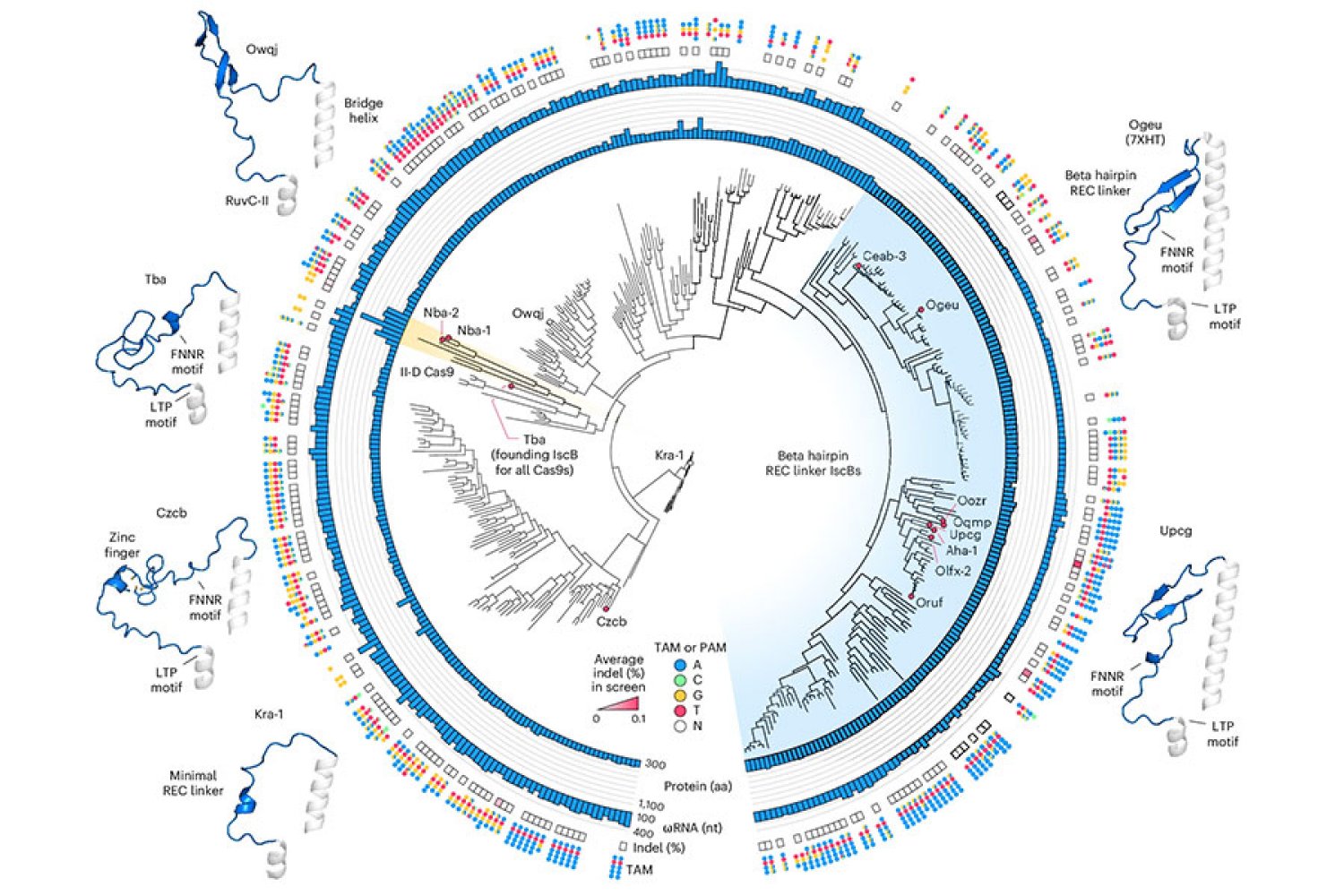
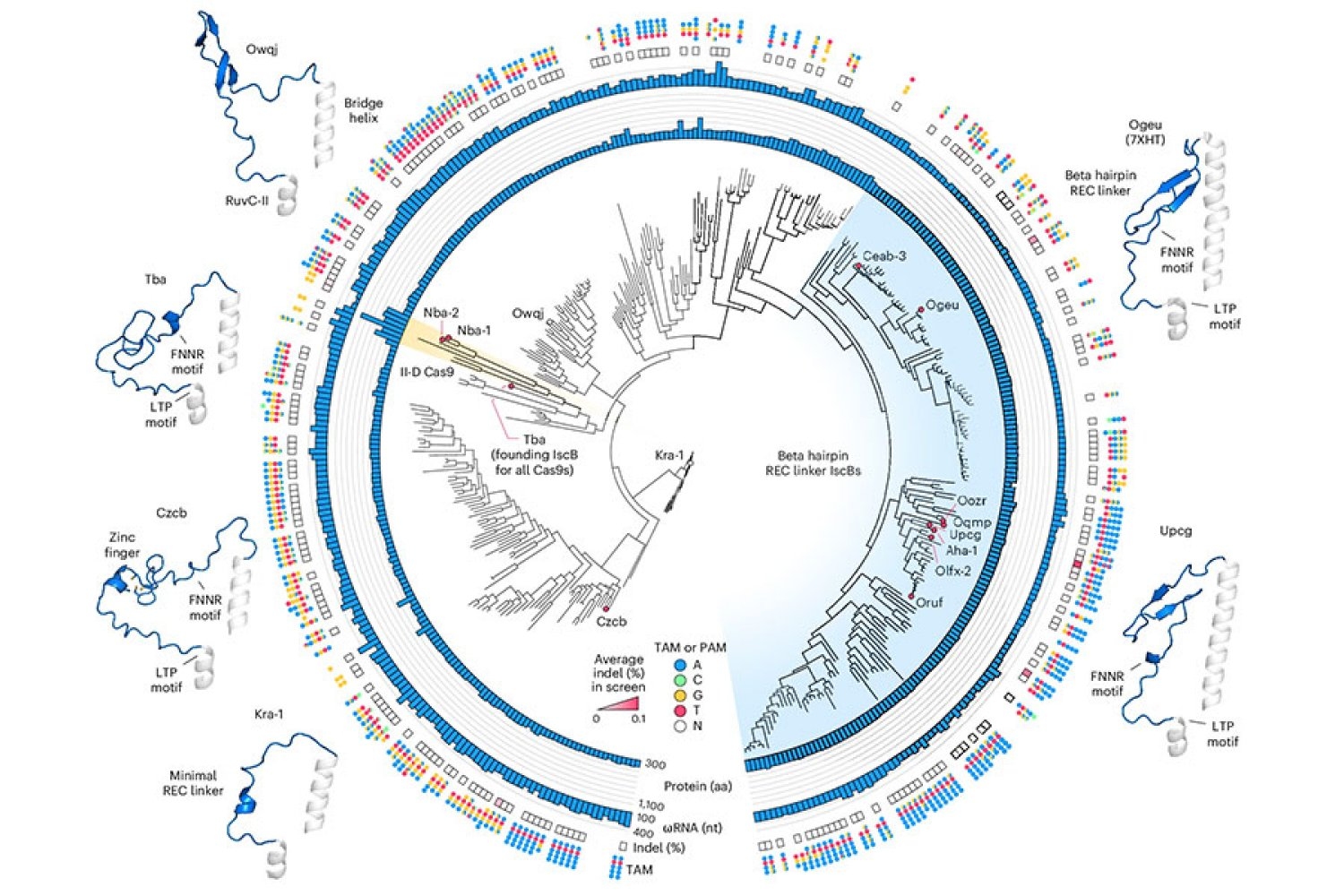
Zu Beginn dieses Ingenieurprozesses, Soumya Kannan, Doktorandin in Zhangs Labor, der jetzt Junior Fellow bei der Harvard Society of Fellows ist, und Postdoc Shiyou Zhu zuerst nach einem ISCB suchte, der einen guten Ausgangspunkt für einen guten Ausgangspunkt ausmacht. Sie testeten quick 400 verschiedene ISCB -Enzyme, die in Bakterien gefunden werden können. Zehn waren in der Lage, DNA in menschlichen Zellen zu bearbeiten.
Selbst die aktivsten derjenigen müssten verbessert werden, um es zu einem nützlichen Genombearbeitungswerkzeug zu machen. Die Herausforderung würde darin bestehen, die Aktivität des Enzyms zu erhöhen, jedoch nur an den Sequenzen, die durch ihre RNA -Anleitung angegeben sind. Wenn das Enzym aktiver würde, aber wahllos, würde es DNA an unbeabsichtigten Stellen schneiden. „Der Schlüssel besteht darin, die Verbesserung der Aktivität und der Spezifität gleichzeitig auszugleichen“, erklärt Zhu.
Zhu merkt an, dass bakterielle ISCBs durch relativ kurze RNA -Guides auf ihre Zielsequenzen gerichtet sind, was es schwierig macht, die Aktivität des Enzyms auf einen bestimmten Teil des Genoms zu beschränken. Wenn ein ISCB so konstruiert werden könnte, dass er einen längeren Leitfaden unterbringt, wäre es weniger wahrscheinlich, dass sie auf Sequenzen über sein beabsichtigtes Ziel hinaus wirken.
Um die ISCB für die Bearbeitung des menschlichen Genoms zu optimieren, nutzte das Workforce Informationen, die der Doktorand Han Altae-tran, der jetzt Postdoc an der College of Washington ist, über die Vielfalt der bakteriellen ISCBs und deren Entwicklung erfahren hatte. Zum Beispiel stellten die Forscher fest, dass ISCBs, die in menschlichen Zellen arbeiteten, ein Phase, das sie als REC bezeichneten, das in anderen ISCBs fehlte. Sie vermuteten, dass das Enzym dieses Phase möglicherweise benötigt, um mit der DNA in menschlichen Zellen zu interagieren. Wenn sie sich die Area genauer ansahen, deutete die Strukturmodellierung darauf hin, dass Rec, indem sie einen leicht expandierenden Teil des Proteins ermöglichen, ISCBS auch längere RNA -Guides erkennen kann.
Basierend auf diesen Beobachtungen experimentierte das Workforce mit dem Austausch in Teilen von Rec -Domänen aus verschiedenen ISCBs und Cas9s und bewertete, wie sich jede Änderung auf die Funktion des Proteins auswirkte. Angeführt von ihrem Verständnis, wie ISCBs und Cas9s sowohl mit DNA als auch mit ihren RNA -Führern interagieren, nahmen die Forscher zusätzliche Änderungen vor und zielten darauf ab, sowohl Effizienz als auch Spezifität zu optimieren.
Am Ende erzeugten sie ein Protein, das sie Novaiscb bezeichneten, das in menschlichen Zellen über 100 -mal aktiver warfare als die ISCB, die sie begonnen hatten, und die eine gute Spezifität für seine Ziele gezeigt hatte.
Kannan und Zhu konstruierten und untersuchten Hunderte neuer ISCBs, bevor sie in Novaiscb ankamen – und jede Veränderung, die sie am ursprünglichen Protein vorgenommen haben, warfare strategisch. Ihre Bemühungen wurden von dem Wissen ihres Groups über die natürliche Entwicklung der ISCBS sowie die Vorhersagen darüber geleitet, wie sich jede Veränderung auf die Struktur des Proteins auswirken würde, die mithilfe eines Instruments für künstliche Intelligenz namens Alphafold2 erstellt wurde. Im Vergleich zu herkömmlichen Methoden zur Einführung zufälliger Veränderungen in ein Protein und Screening für ihre Auswirkungen beschleunigte dieser rationale Engineering -Ansatz die Fähigkeit des Groups, ein Protein mit den von ihnen gesuchten Merkmalen zu identifizieren.
Das Workforce hat gezeigt, dass Novaiscb ein gutes Gerüst für eine Vielzahl von Genombearbeitungswerkzeugen ist. „Es funktioniert biochemisch sehr ähnlich wie Cas9, und das macht es einfach, über Werkzeuge zu portieren, die bereits mit dem Cas9 -Gerüst optimiert wurden“, sagt Kannan. Bei unterschiedlichen Modifikationen verwendeten die Forscher NovaISCB, um spezifische Buchstaben des DNA -Code in menschlichen Zellen zu ersetzen und die Aktivität gezielter Gene zu ändern.
Wichtig ist, dass die Instruments auf der Foundation von NovaiscB so kompakt genug sind, um in einem einzelnen Adeno-assoziierten Virus (AAV) leicht verpackt zu werden-der Vektor, der am häufigsten verwendet wird, um Patienten sicher die Gentherapie zu liefern. Da sie sperriger sind, können Instruments, die mit Cas9 entwickelt wurden, eine kompliziertere Bereitstellungsstrategie erfordern.
Das Workforce von Novaiscb nach therapeutischem Gebrauch erstellte ein Device namens Omegaoff, das der DNA chemische Marker hinzufügt, um die Aktivität bestimmter Gene abzuwählen. Sie programmierten Omegaoff, um ein Gen zu unterdrücken, das an der Cholesterinregulation beteiligt ist, und verwendeten dann AAV, um das System an die Lebern von Mäusen zu liefern, was zu einer dauerhaften Verringerung der Cholesterinspiegel im Blut der Tiere führte.
Das Workforce geht davon aus, dass NovaISCB verwendet werden kann, um Genombearbeitungswerkzeuge für die meisten menschlichen Gene anzusprechen, und freuen sich darauf, zu sehen, wie andere Labors die neue Technologie einsetzen. Sie hoffen auch, dass andere ihren evolutiongesteuerten Ansatz zur rationalen Proteintechnik übernehmen werden. „Die Natur hat eine solche Vielfalt und ihre Systeme haben unterschiedliche Vor- und Nachteile“, sagt Zhu. „Indem wir diese natürliche Vielfalt kennenlernen, können wir die Systeme, die wir versuchen, immer besser zu konstruieren.“
Diese Studie wurde zum Teil vom Okay. Lisa Yang und dem Hock E. Tan Heart for Molekular Therapeutics am MIT, dem breiten Institut für programmierbare Therapeutika -Geschenkspender, Pershing Sq. Basis, William Ackman, Neri Oxman, The Phillips Household und J. und P. Poitras finanziert.