Da die Fähigkeiten generativer KI-Modelle gewachsen sind, haben Sie wahrscheinlich gesehen, wie sie einfache Textaufforderungen in hyperrealistische Bilder und sogar erweiterte Videoclips umwandeln können.
In jüngerer Zeit hat die generative KI gezeigt, dass sie Chemikern und Biologen dabei helfen kann, statische Moleküle wie Proteine und DNA zu erforschen. Modelle wie AlphaFold können molekulare Strukturen vorhersagen, um die Entdeckung von Arzneimitteln zu beschleunigen, und das vom MIT unterstützte „HF-Diffusion„kann beispielsweise dabei helfen, neue Proteine zu entwickeln. Eine Herausforderung besteht jedoch darin, dass sich Moleküle ständig bewegen und hin und her bewegen, was bei der Konstruktion neuer Proteine und Medikamente wichtig ist. Die Simulation dieser Bewegungen auf einem Laptop mithilfe der Physik – einer Technik, die als Molekulardynamik bekannt ist – kann sehr teuer sein und auf Supercomputern Milliarden von Zeitschritten erfordern.
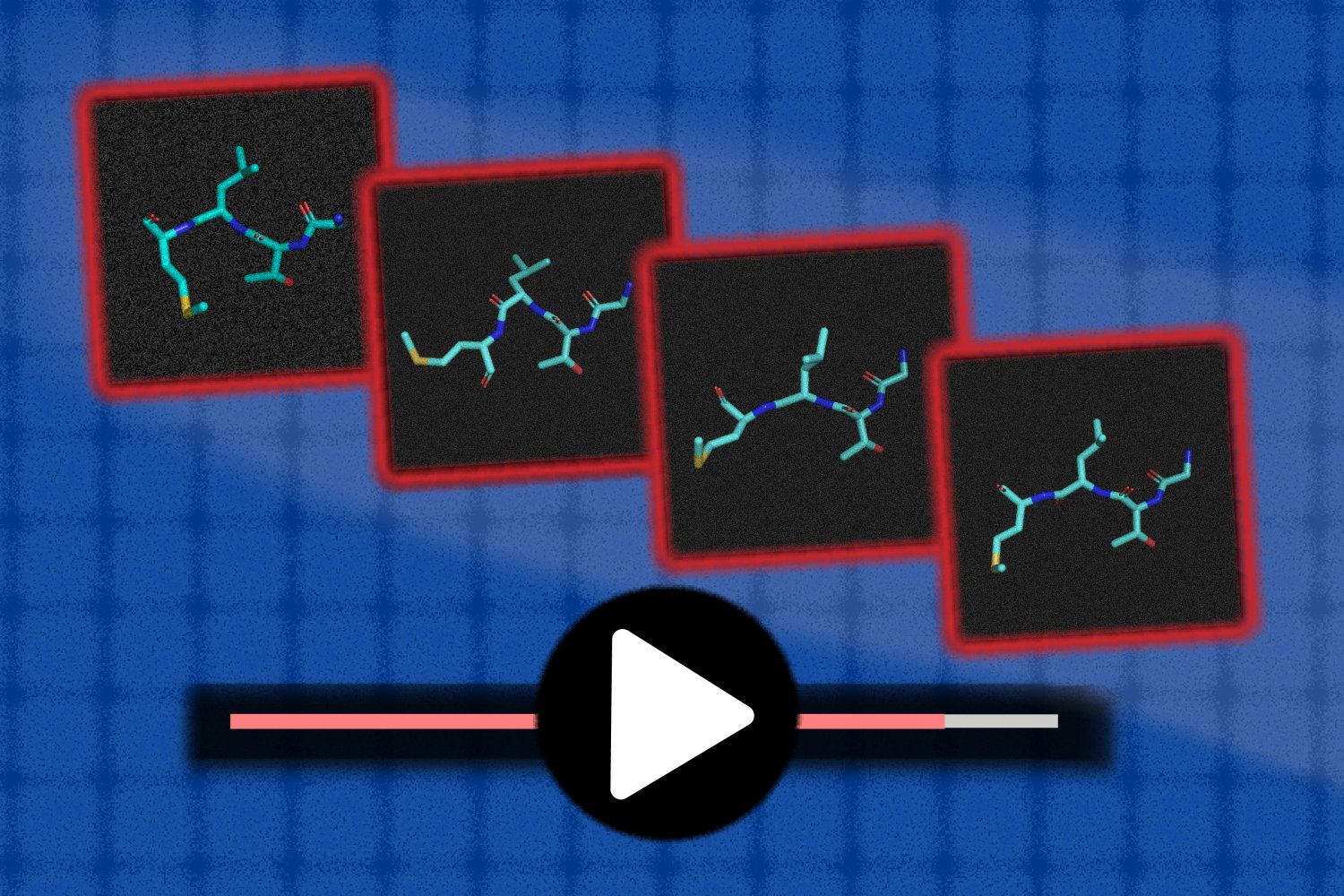
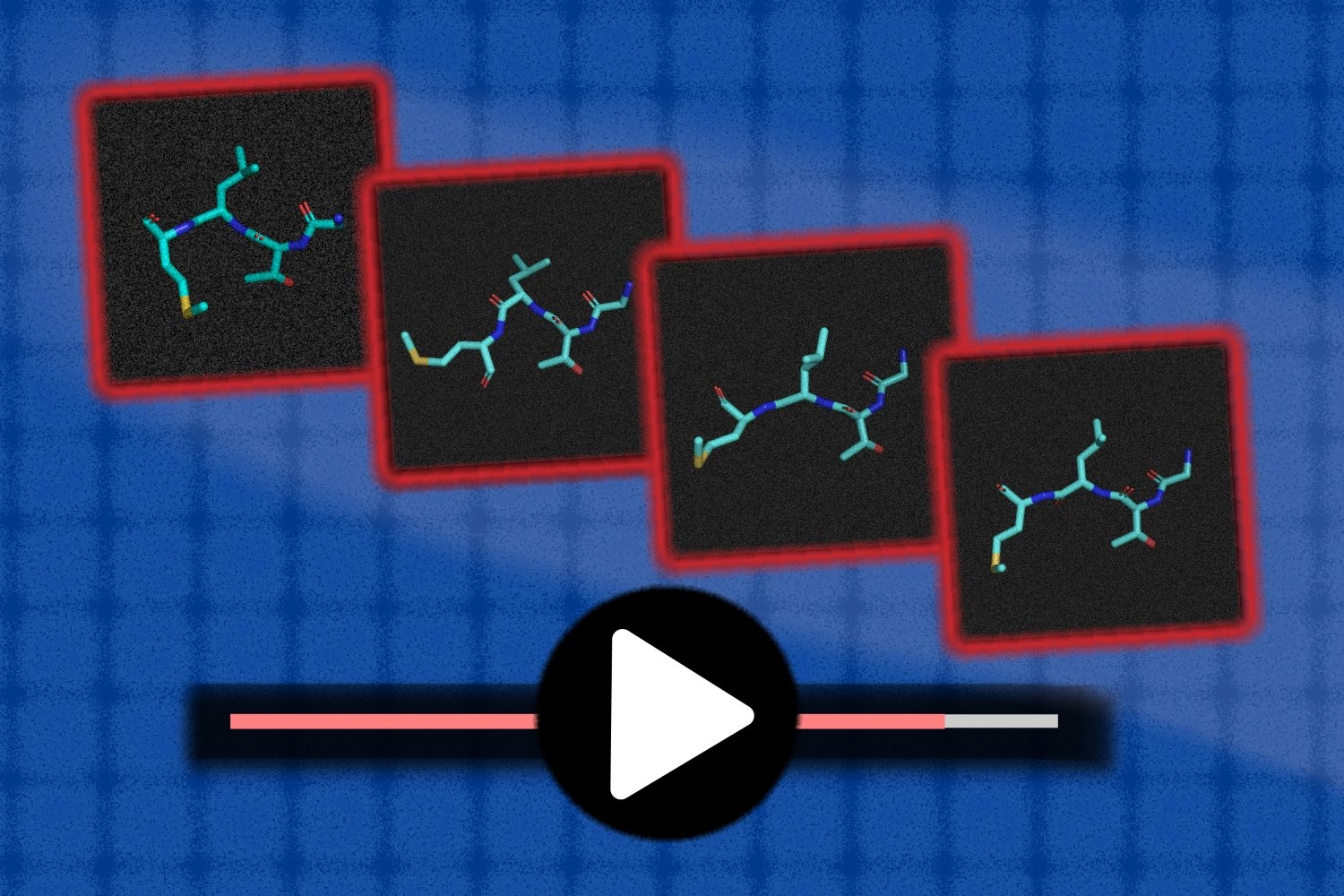
Um diese Verhaltensweisen effizienter zu simulieren, haben Forscher des MIT Laptop Science and Synthetic Intelligence Laboratory (CSAIL) und der Fakultät für Mathematik ein generatives Modell entwickelt, das aus früheren Daten lernt. Das System des Groups namens MDGen kann ein Bild eines 3D-Moleküls aufnehmen und wie in einem Video simulieren, was als nächstes passieren wird, separate Standbilder verbinden und sogar fehlende Bilder ergänzen. Durch Drücken der „Play-Style“ für Moleküle könnte das Instrument möglicherweise Chemikern dabei helfen, neue Moleküle zu entwerfen und genau zu untersuchen, wie intestine ihre Medikamentenprototypen für Krebs und andere Krankheiten mit der Molekülstruktur, die sie beeinflussen möchten, interagieren würden.
Co-Hauptautor Bowen Jing SM ’22 sagt, dass MDGen ein erster Proof of Idea ist, aber es deutet auf den Beginn einer aufregenden neuen Forschungsrichtung hin. „Schon früh erzeugten generative KI-Modelle eher einfache Movies, etwa das Blinzeln einer Individual oder das Wedeln eines Hundes mit dem Schwanz“, sagt Jing, Doktorandin am CSAIL. „Ein paar Jahre später haben wir erstaunliche Modelle wie Sora oder Veo, die auf viele interessante Arten nützlich sein können. Wir hoffen, eine ähnliche Imaginative and prescient für die molekulare Welt zu vermitteln, in der dynamische Trajektorien die Movies sind. Sie können dem Modell beispielsweise das erste und das zehnte Bild geben und es animiert, was dazwischen liegt, oder es kann Rauschen aus einem molekularen Video entfernen und erraten, was verborgen battle.“
Die Forscher sagen, dass MDGen einen Paradigmenwechsel gegenüber früheren vergleichbaren Arbeiten mit generativer KI in einer Weise darstellt, die viel breitere Anwendungsfälle ermöglicht. Frühere Ansätze waren „autoregressiv“, das heißt, sie stützten sich bei der Erstellung des nächsten auf das vorherige Standbild und begannen beim Erstellen einer Videosequenz mit dem allerersten Bild. Im Gegensatz dazu generiert MDGen die Frames parallel zur Diffusion. Dies bedeutet, dass MDGen beispielsweise zum Verbinden von Frames an den Endpunkten oder zum „Upsampling“ einer Trajektorie mit niedriger Framerate zusätzlich zum Drücken der Play-Style beim ersten Body verwendet werden kann.
Diese Arbeit wurde in einem Papier vorgestellt, das auf der Convention on Neural Info Processing Programs (NeurIPS) im vergangenen Dezember gezeigt wurde. Letzten Sommer wurde es für seine potenziellen kommerziellen Auswirkungen auf dem ML4LMS-Workshop der Worldwide Convention on Machine Studying ausgezeichnet.
Einige kleine Fortschritte für die Molekulardynamik
In Experimenten stellten Jing und seine Kollegen fest, dass die Simulationen von MDGen der direkten Ausführung physikalischer Simulationen ähnelten, Trajektorien jedoch 10 bis 100 Mal schneller erzeugten.
Das Staff testete zunächst die Fähigkeit seines Modells, einen 3D-Rahmen eines Moleküls aufzunehmen und die nächsten 100 Nanosekunden zu erzeugen. Ihr System setzte aufeinanderfolgende 10-Nanosekunden-Blöcke zusammen, damit diese Generationen diese Dauer erreichen konnten. Das Staff stellte fest, dass MDGen mit der Genauigkeit eines Basismodells mithalten konnte und den Videogenerierungsprozess in etwa einer Minute abschloss – ein Bruchteil der drei Stunden, die das Basismodell zur Simulation derselben Dynamik benötigte.
Als MDGen das erste und letzte Bild einer Ein-Nanosekunden-Sequenz erhielt, modellierte es auch die Schritte dazwischen. Das System der Forscher zeigte bei über 100.000 verschiedenen Vorhersagen ein gewisses Maß an Realismus: Es simulierte wahrscheinlichere molekulare Flugbahnen als seine Basislinien auf Clips, die kürzer als 100 Nanosekunden waren. In diesen Assessments zeigte MDGen auch die Fähigkeit, Peptide zu verallgemeinern, die es zuvor nicht gesehen hatte.
Zu den Fähigkeiten von MDGen gehört auch die Simulation von Frames innerhalb von Frames und das „Upsampling“ der Schritte zwischen jeder Nanosekunde, um schnellere molekulare Phänomene besser zu erfassen. Es kann sogar Strukturen von Molekülen „einfärben“ und so entfernte Informationen über sie wiederherstellen. Diese Merkmale könnten schließlich von Forschern genutzt werden, um Proteine auf der Grundlage einer Spezifikation zu entwerfen, wie sich verschiedene Teile des Moleküls bewegen sollen.
Mit der Proteindynamik herumspielen
Jing und Co-Hauptautor Hannes Stärk sagen, dass MDGen ein frühes Zeichen für Fortschritte bei der effizienteren Erzeugung der Molekulardynamik ist. Dennoch fehlen ihnen die Daten, um diese Modelle unmittelbar für die Entwicklung von Arzneimitteln oder Molekülen nutzbar zu machen, die die Bewegungen auslösen, die Chemiker in einer Zielstruktur sehen möchten.
Die Forscher wollen MDGen von der Modellierung von Molekülen bis hin zur Vorhersage, wie sich Proteine im Laufe der Zeit verändern, skalieren. „Derzeit nutzen wir Spielzeugsysteme“, sagt Stärk, ebenfalls Doktorand am CSAIL. „Um die Vorhersagefähigkeiten von MDGen zur Modellierung von Proteinen zu verbessern, müssen wir auf der aktuellen Architektur und den verfügbaren Daten aufbauen. Wir verfügen noch nicht über ein Repository im YouTube-Maßstab für diese Artwork von Simulationen, daher hoffen wir, eine separate Methode des maschinellen Lernens zu entwickeln, die den Datenerfassungsprozess für unser Modell beschleunigen kann.“
Derzeit stellt MDGen einen ermutigenden Weg nach vorn bei der Modellierung molekularer Veränderungen dar, die für das bloße Auge unsichtbar sind. Chemiker könnten diese Simulationen auch nutzen, um tiefer in das Verhalten von Medikamentenprototypen für Krankheiten wie Krebs oder Tuberkulose einzutauchen.
„Maschinelle Lernmethoden, die aus physikalischen Simulationen lernen, stellen eine aufkeimende neue Grenze in der KI für die Wissenschaft dar“, sagt Bonnie Berger, Professorin für Mathematik am MIT Simons, CSAIL-Hauptforscherin und leitende Autorin des Papiers. „MDGen ist ein vielseitiges, vielseitiges Modellierungsframework, das diese beiden Bereiche verbindet, und wir freuen uns sehr, unsere frühen Modelle in diese Richtung zu teilen.“
„Die Probenahme realistischer Übergangspfade zwischen molekularen Zuständen ist eine große Herausforderung“, sagt Tommi Jaakkola, Mitautor und leitender Autor. Er ist Thomas-Siebel-Professor für Elektrotechnik und Informatik am MIT und am Institute for Knowledge, Programs, and Society sowie CSAIL-Hauptforscher . „Diese frühe Arbeit zeigt, wie wir beginnen könnten, solche Herausforderungen anzugehen, indem wir die generative Modellierung auf vollständige Simulationsläufe verlagern.“
Forscher aus dem gesamten Bereich der Bioinformatik haben dieses System für seine Fähigkeit gepriesen, molekulare Transformationen zu simulieren. „MDGen modelliert Molekulardynamiksimulationen als eine gemeinsame Verteilung struktureller Einbettungen und erfasst molekulare Bewegungen zwischen diskreten Zeitschritten“, sagt Simon Olsson, außerordentlicher Professor der Chalmers College of Know-how, der nicht an der Forschung beteiligt battle. „MDGen nutzt ein maskiertes Lernziel und ermöglicht modern Anwendungsfälle wie das Sampling von Übergangspfaden und das Zeichnen von Analogien zum Inpainting von Trajektorien, die metastabile Phasen verbinden.“
Die Arbeit der Forscher an MDGen wurde teilweise vom Nationwide Institute of Normal Medical Sciences, dem US-Energieministerium, der Nationwide Science Basis, dem Machine Studying for Pharmaceutical Discovery and Synthesis Consortium und der Abdul Latif Jameel Clinic for Machine Studying unterstützt in Well being, der Protection Risk Discount Company und der Protection Superior Analysis Initiatives Company.