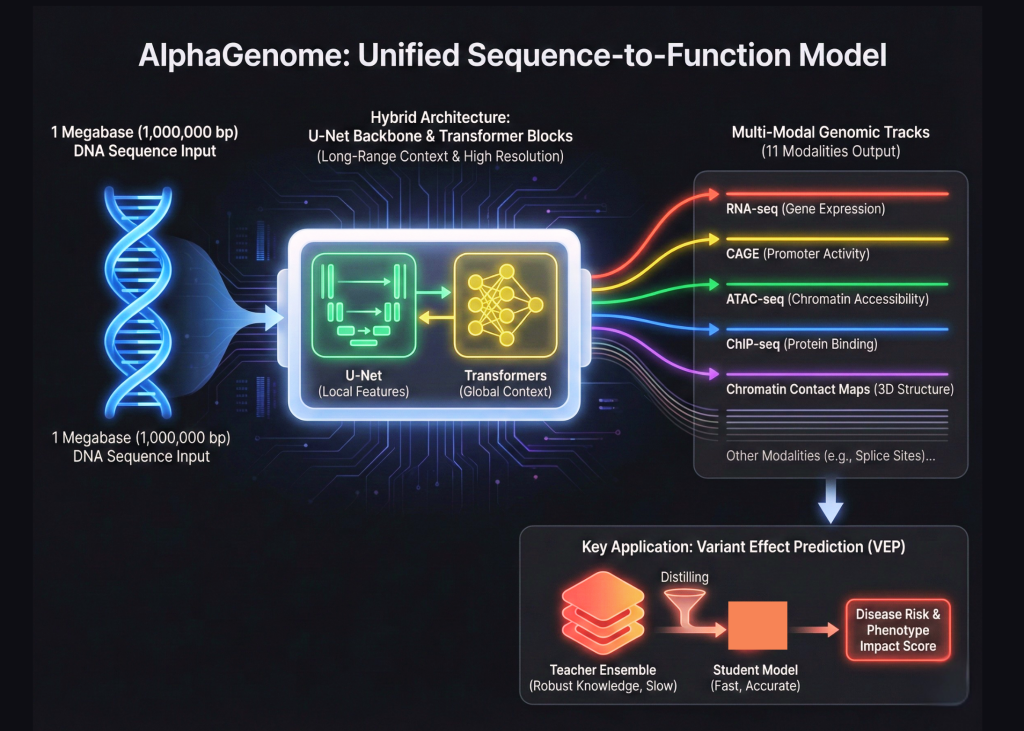
Google DeepMind erweitert sein biologisches Toolkit über die Welt der Proteinfaltung hinaus. Nach dem Erfolg von AlphaFold hat das Forschungsteam von Google AlphaGenome eingeführt. Hierbei handelt es sich um ein einheitliches Deep-Studying-Modell, das für die Sequenz-zu-Funktions-Genomik entwickelt wurde. Dies stellt einen großen Wandel in der Artwork und Weise dar, wie wir das menschliche Genom modellieren. AlphaGenome behandelt DNA nicht als einfachen Textual content. Stattdessen verarbeitet es 1.000.000 Basenpaarfenster roher DNA, um den Funktionszustand einer Zelle vorherzusagen.
Überbrückung der Skalenlücke mit Hybridarchitekturen
Die Komplexität des menschlichen Genoms ergibt sich aus seiner Größe. Den meisten bestehenden Modellen fällt es schwer, das Gesamtbild zu erkennen und gleichzeitig die feinen Particulars im Auge zu behalten. AlphaGenome löst dieses Drawback durch den Einsatz einer Hybridarchitektur. Es kombiniert ein U-Internet-Spine mit Transformer-Blöcken. Dadurch kann das Modell weitreichende Wechselwirkungen über eine Sequenz von 1 Megabase erfassen und gleichzeitig die Basenpaarauflösung beibehalten. Das ist, als würde man ein System aufbauen, das ein tausendseitiges Buch lesen kann und sich trotzdem die genaue Place eines einzelnen Kommas merkt.
Zuordnung von Sequenzen zu funktionellen biologischen Modalitäten
AlphaGenome ist ein Sequenz-zu-Funktions-Modell. Das bedeutet, dass sein Hauptziel darin besteht, DNA-Sequenzen direkt biologischen Aktivitäten zuzuordnen. Diese Aktivitäten werden in genomischen Spuren gemessen. Das Forschungsteam trainierte AlphaGenome, 11 verschiedene genomische Modalitäten vorherzusagen. Zu diesen Modalitäten gehören RNA-seq, CAGE und ATAC-seq. Dazu gehören auch ChIP-seq für verschiedene Transkriptionsfaktoren und Chromatin-Kontaktkarten. Durch die gleichzeitige Vorhersage all dieser Spuren erhält das Modell ein ganzheitliches Verständnis darüber, wie DNA die Zelle reguliert.
Die Kraft des Multitasking-Lernens in der Genomik
Der technische Fortschritt von AlphaGenome liegt in seiner Fähigkeit, 11 verschiedene Datentypen gleichzeitig zu verarbeiten. In der Vergangenheit haben Forscher oft für jede Aufgabe separate Modelle erstellt. AlphaGenome verwendet einen Multitasking-Lernansatz. Dies hilft dem Modell, gemeinsame Merkmale verschiedener biologischer Prozesse zu lernen. Wenn das Modell versteht, wie ein Protein an DNA bindet, kann es besser vorhersagen, wie diese DNA als RNA ausgedrückt wird. Dieser einheitliche Ansatz reduziert den Bedarf an mehreren Spezialmodellen.
Verbesserung der Vorhersage von Varianteneffekten durch Destillation
Eine der wichtigsten Anwendungen für AlphaGenome ist die Variant Impact Prediction (VEP). Dieser Prozess bestimmt, wie sich eine einzelne Mutation in der DNA auf den Körper auswirkt. Mutationen können zu Krankheiten wie Krebs oder Herzerkrankungen führen. AlphaGenome zeichnet sich dabei durch die Verwendung einer speziellen Trainingsmethode aus, die „Lehrer-Schüler-Destillation“ genannt wird. Das Forschungsteam erstellte zunächst ein Ensemble von „All-Folds“-Lehrermodellen. Diese Lehrer wurden anhand riesiger Mengen genomischer Daten geschult. Anschließend destillierten sie dieses Wissen in einem einzigen Studentenmodell.
Komprimiertes Wissen für die Präzisionsmedizin
Dieser Destillationsprozess macht das Modell sowohl schneller als auch robuster. Dies ist eine Standardmethode zur Komprimierung von Wissen. Die Anwendung auf die Genomik in diesem Maßstab ist jedoch ein neuer Meilenstein. Das Schülermodell lernt, die qualitativ hochwertigen Vorhersagen des Lehrerensembles zu reproduzieren. Dadurch können schädliche Mutationen mit hoher Genauigkeit identifiziert werden. Das Modell kann sogar vorhersagen, wie sich eine Mutation in einem entfernten regulatorischen Component auf ein weit entferntes Gen auf dem DNA-Strang auswirken könnte.
Hochleistungsrechnen mit JAX und TPUs
Die Architektur wird mit JAX implementiert. JAX ist eine leistungsstarke numerische Computerbibliothek. Es wird bei Google häufig für groß angelegtes maschinelles Lernen verwendet. Durch die Verwendung von JAX kann AlphaGenome effizient auf Tensor Processing Models oder TPUs ausgeführt werden. Das Forschungsteam nutzte Sequenzparallelität, um die riesigen Eingabefenster von 1 Megabase zu bewältigen. Dadurch wird sichergestellt, dass der Speicherbedarf mit zunehmender Sequenzlänge nicht explodiert. Dies zeigt, wie wichtig es ist, den richtigen Rahmen für groß angelegte biologische Daten auszuwählen.
Transferlernen für datenarme Zelltypen
AlphaGenome befasst sich auch mit der Herausforderung der Datenknappheit bei bestimmten Zelltypen. Da es sich um ein Basismodell handelt, kann es für bestimmte Aufgaben fein abgestimmt werden. Das Modell lernt allgemeine biologische Regeln aus großen öffentlichen Datensätzen. Diese Regeln können dann auf seltene Krankheiten oder bestimmte Gewebe angewendet werden, für die Daten schwer zu finden sind. Diese Fähigkeit zum Transferlernen ist einer der Gründe, warum AlphaGenome so vielseitig ist. Es kann vorhersagen, wie sich ein Gen in einer Gehirnzelle verhält, selbst wenn es hauptsächlich anhand von Leberzelldaten trainiert wurde.
Auf dem Weg zu einer neuen Ära der personalisierten Pflege
In Zukunft könnte AlphaGenome eine neue Ära der personalisierten Medizin einläuten. Ärzte könnten das Modell verwenden, um das gesamte Genom eines Patienten in 1.000.000 Basenpaar-Blöcken zu scannen. Sie könnten genau identifizieren, welche Varianten wahrscheinlich gesundheitliche Probleme verursachen. Dies würde Behandlungen ermöglichen, die auf den spezifischen genetischen Code einer Individual zugeschnitten sind. AlphaGenome bringt uns dieser Realität näher, indem es eine klare und genaue Karte des funktionellen Genoms liefert.
Den Customary für biologische KI setzen
AlphaGenome markiert auch einen Wendepunkt für KI in der Genomik. Es beweist, dass wir die komplexesten biologischen Systeme mithilfe derselben Prinzipien modellieren können, die auch in der modernen KI verwendet werden. Durch die Kombination von U-Internet-Strukturen mit Transformers und der Verwendung der Lehrer-Schüler-Destillation hat das Google DeepMind-Staff einen neuen Customary gesetzt.
Wichtige Erkenntnisse
- Hybride Sequenzarchitektur: AlphaGenome verwendet ein spezielles Hybriddesign, das Folgendes kombiniert: U-Internet Rückgrat mit Transformator Blöcke. Dadurch kann das Modell große Fenster verarbeiten 1.000.000 Basenpaare unter Beibehaltung der hohen Auflösung, die zur Identifizierung einzelner Mutationen erforderlich ist.
- Multimodale funktionale Vorhersage: Das Modell ist darauf trainiert, Vorhersagen zu treffen 11 verschiedene genomische Modalitäten gleichzeitig, einschließlich RNA-seq, CAGE und ATAC-seq. Durch das gemeinsame Erlernen dieser verschiedenen biologischen Spuren erhält das System ein ganzheitliches Verständnis darüber, wie DNA die Zellaktivität in verschiedenen Geweben reguliert.
- Lehrer-Schüler-Destillation: Um branchenführende Genauigkeit zu erreichen Varianteneffektvorhersage (VEP)Forscher verwendeten eine Destillationsmethode. Sie übertrugen das Wissen aus einem Ensemble leistungsstarker „Lehrer“-Modelle in ein einziges, effizientes „Schüler“-Modell, das schneller und robuster bei der Identifizierung krankheitsverursachender Mutationen ist.
- Gebaut für Hochleistungsrechnen: Das Framework ist implementiert in JAX und optimiert für TPUs. Durch die Verwendung von Sequenzparallelität kann AlphaGenome die Rechenlast der Analyse von DNA-Sequenzen im Megabasen-Maßstab bewältigen, ohne die Speichergrenzen zu überschreiten, was es zu einem leistungsstarken Werkzeug für groß angelegte Forschung macht.
Schauen Sie sich das an Papier Und Repo. Sie können uns auch gerne weiter folgen Twitter und vergessen Sie nicht, bei uns mitzumachen 100.000+ ML SubReddit und Abonnieren Unser Publication. Warten! Bist du im Telegram? Jetzt können Sie uns auch per Telegram kontaktieren.