Ein Group von Forschern der Stanford Drugs hat SleepFM Scientific eingeführt, ein multimodales Schlafgrundlagenmodell, das aus der klinischen Polysomnographie lernt und das langfristige Krankheitsrisiko anhand einer einzigen Nacht Schlaf vorhersagt. Die Forschungsarbeit wurde in Nature Drugs veröffentlicht und das Group hat den klinischen Code als Open Supply veröffentlicht sleepfm-clinical Repository auf GitHub unter der MIT-Lizenz.
Von der Nacht-Polysomnographie bis zur allgemeinen Darstellung
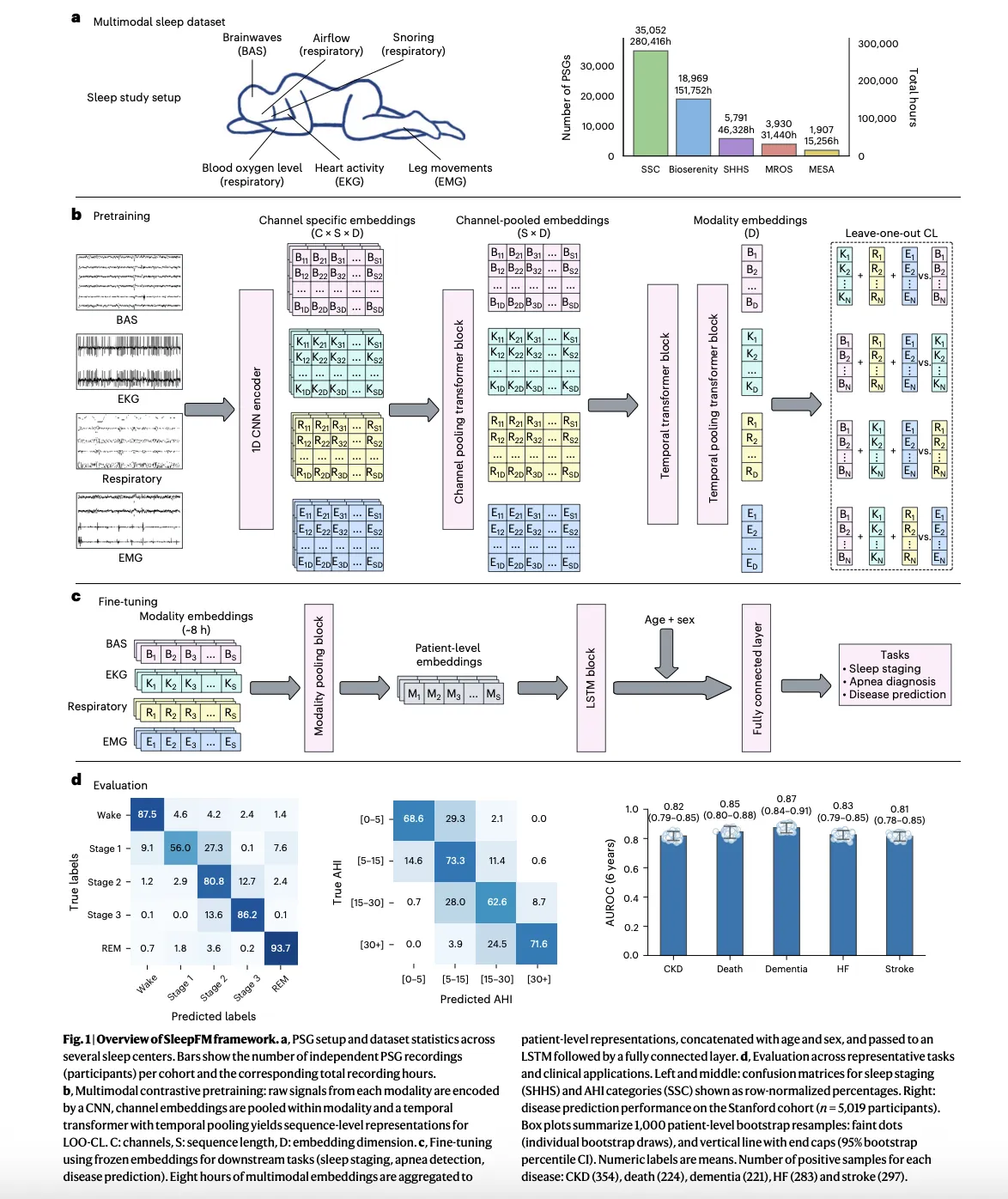
Die Polysomnographie zeichnet während einer ganzen Nacht in einem Schlaflabor Gehirnaktivität, Augenbewegungen, Herzsignale, Muskeltonus, Atemanstrengung und Sauerstoffsättigung auf. Es handelt sich um den Goldstandard-Take a look at in der Schlafmedizin, aber die meisten klinischen Arbeitsabläufe verwenden ihn nur für die Schlafstadienbestimmung und die Schlafapnoe-Diagnose. Das Forschungsteam behandelt diese Mehrkanalsignale als dichte physiologische Zeitreihen und trainiert ein Basismodell, um eine gemeinsame Darstellung über alle Modalitäten hinweg zu erlernen.
SleepFM basiert auf etwa 585.000 Stunden Schlafaufzeichnungen von etwa 65.000 Menschen aus mehreren Kohorten. Die größte Kohorte stammt vom Stanford Sleep Drugs Heart, wo zwischen 1999 und 2024 etwa 35.000 Erwachsene und Kinder über Nacht Studien absolvierten. Diese klinische Kohorte ist mit elektronischen Gesundheitsakten verknüpft, die später eine Überlebensanalyse für Hunderte von Krankheitskategorien ermöglichen.
Modellarchitektur und Vortrainingsziel
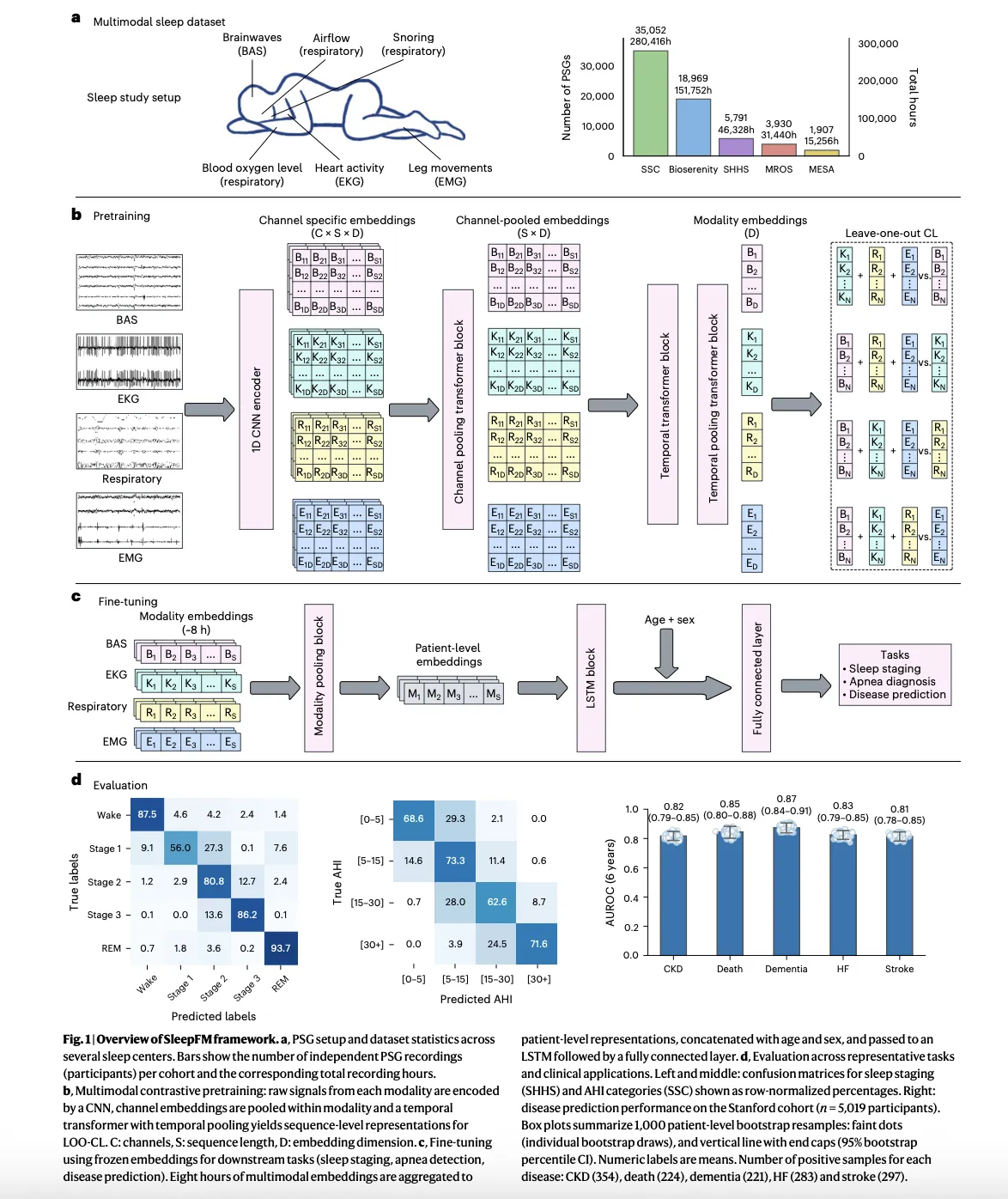
Auf der Modellierungsebene verwendet SleepFM ein Faltungs-Spine, um lokale Merkmale aus jedem Kanal zu extrahieren, gefolgt von einer aufmerksamkeitsbasierten Aggregation über Kanäle hinweg und einem zeitlichen Transformator, der über kurze Abschnitte der Nacht arbeitet. Die gleiche Kernarchitektur tauchte bereits in früheren Arbeiten zu SleepFM zur Schlafstadieneinstufung und zur Erkennung von Atemstörungen im Schlaf auf, wo gezeigt wurde, dass das Erlernen der gemeinsamen Einbettung von Gehirnaktivität, Elektrokardiographie und Atmungssignalen die nachgelagerte Leistung verbessert.
Das Ziel vor dem Coaching besteht darin, kontrastives Lernen auszulassen. Für jedes kurze Zeitsegment erstellt das Modell separate Einbettungen für jede Modalitätsgruppe, wie z. B. Gehirnsignale, Herzsignale und Atmungssignale, und lernt dann, diese Modalitätseinbettungen so auszurichten, dass jede Teilmenge die gemeinsame Darstellung der verbleibenden Modalitäten vorhersagt. Dieser Ansatz macht das Modell strong gegenüber fehlenden Kanälen und heterogenen Aufnahmemontagen, die in realen Schlaflaboren häufig vorkommen.
Nach dem Vortraining zur unmarkierten Polysomnographie wird das Rückgrat eingefroren und kleine aufgabenspezifische Köpfe trainiert. Für Normal-Schlafaufgaben ordnet ein leichter rezidivierender oder linearer Kopf Einbettungen den Schlafstadien oder Apnoe-Bezeichnungen zu. Zur Vorhersage des klinischen Risikos aggregiert das Modell die gesamte Nacht in einer einzigen Einbettung auf Patientenebene, verkettet grundlegende demografische Daten wie Alter und Geschlecht und speist diese Darstellung dann in eine Cox-Proportional-Hazards-Schicht zur Modellierung der Zeit bis zum Ereignis ein.
Benchmarks zu Schlafstadien und Apnoe
Bevor es zur Krankheitsvorhersage überging, überprüfte das Forschungsteam, dass SleepFM bei Standardaufgaben der Schlafanalyse mit Spezialmodellen konkurriert. Frühere Arbeiten haben bereits gezeigt, dass ein einfacher Klassifikator Zusätzlich zu den SleepFM-Einbettungen übertreffen sie Finish-to-Finish-Faltungsnetzwerke bei der Klassifizierung von Schlafstadien und bei der Erkennung schlafbezogener Atmungsstörungen, mit Zuwächsen bei Makro-AUROC und AUPRC bei mehreren öffentlichen Datensätzen.
In der klinischen Studie wird dasselbe vortrainierte Rückgrat für die Schlafeinteilung und die Klassifizierung des Apnoe-Schweregrads in multizentrischen Kohorten wiederverwendet. Die in der Forschungsarbeit berichteten Ergebnisse zeigen, dass SleepFM bestehenden Instruments wie traditionellen Faltungsmodellen und anderen automatisierten Schlaf-Staging-Systemen entspricht oder diese übertrifft, was bestätigt, dass die Darstellung die Kernschlafphysiologie und nicht nur statistische Artefakte aus einem einzelnen Datensatz erfasst.
Vorhersage von 130 Krankheiten und Todesfällen aus einer Nacht Schlaf
Der Kernbeitrag dieser Stanford-Forschungsarbeit ist die Krankheitsvorhersage. Das Forschungsteam ordnet Diagnosecodes in den elektronischen Gesundheitsakten von Stanford Phäcodes zu und definiert mehr als 1.000 mögliche Krankheitsgruppierungen. Für jeden Phecode berechnen sie die Zeit bis zur ersten Diagnose nach der Schlafstudie und passen ein Cox-Modell an die SleepFM-Einbettungen an.
SleepFM identifiziert 130 Krankheitsausgänge, deren Risiken anhand einer einzigen Polysomnographienacht mit starker Unterscheidung vorhersehbar sind. Dazu gehören alle Ursachen für Mortalität, Demenz, Myokardinfarkt, Herzinsuffizienz, chronische Nierenerkrankung, Schlaganfall, Vorhofflimmern, verschiedene Krebsarten sowie zahlreiche psychiatrische und metabolische Störungen. Für viele dieser Erkrankungen liegen Leistungsmetriken wie der Konkordanzindex und die Fläche unter der Empfängerbetriebskurve in Bereichen, die mit etablierten Risikobewertungen vergleichbar sind, auch wenn das Modell nur Schlafaufzeichnungen plus grundlegende demografische Daten verwendet.
In der Berichterstattung wird außerdem darauf hingewiesen, dass die auf SleepFM basierenden Vorhersagen für einige Krebsarten, Schwangerschaftskomplikationen, Kreislauferkrankungen und psychische Störungen eine Genauigkeit von etwa 80 Prozent für mehrjährige Risikofenster erreichen. Dies deutet darauf hin, dass subtile Muster in der Koordination zwischen Gehirn-, Herz- und Atemsignalen Informationen über latente Krankheitsprozesse enthalten, die noch nicht klinisch sichtbar sind.
Vergleich mit einfacheren Basislinien
Um den Mehrwert zu bewerten, verglich das Forschungsteam SleepFM-basierte Risikomodelle mit zwei Basislinien. Die erste verwendet nur demografische Merkmale wie Alter, Geschlecht und Physique-Mass-Index. Der zweite Schritt trainiert ein Finish-to-Finish-Modell direkt auf der Grundlage der Polysomnographie und der Ergebnisse, ohne unbeaufsichtigtes Vortraining. In den meisten Krankheitskategorien führt die vorab trainierte SleepFM-Darstellung in Kombination mit einem einfachen Überlebenskopf zu einer höheren Konkordanz und einem höheren AUROC für den langen Horizont als beide Basislinien.
Diese Forschung zeigt deutlich, dass der Gewinn weniger von einem komplexen Vorhersagekopf als vielmehr vom Grundlagenmodell herrührt, das eine allgemeine Darstellung der Schlafphysiologie gelernt hat. In der Praxis bedeutet dies, dass klinische Zentren ein einziges vorab trainiertes Grundgerüst wiederverwenden, kleine ortsspezifische Köpfe mit relativ bescheidenen gekennzeichneten Kohorten erlernen und dennoch die Leistung auf dem neuesten Stand erreichen können.
Schauen Sie sich das an Papier Und VOLLSTÄNDIGE CODES hier. Sie können uns auch gerne weiter folgen Twitter und vergessen Sie nicht, bei uns mitzumachen 100.000+ ML SubReddit und Abonnieren Unser E-newsletter. Warten! Bist du im Telegram? Jetzt können Sie uns auch per Telegram kontaktieren.
Schauen Sie sich unsere neueste Model von anai2025.deveine auf das Jahr 2025 ausgerichtete Analyseplattform, die Modelleinführungen, Benchmarks und Ökosystemaktivitäten in einen strukturierten Datensatz umwandelt, den Sie filtern, vergleichen und exportieren können
Der Beitrag Stanford-Forscher entwickeln SleepFM Scientific: Ein multimodales KI-Modell der Sleep Basis für die Vorhersage von Krankheiten über 130 erschien zuerst auf MarkTechPost.