Es besteht weltweit ein Mangel an Zugang zu Dolmetschern durch medizinische Bildgebungsexperten in allen Fachgebieten, einschließlich Radiologie, Dermatologie Und Pathologie. Die Technologie des maschinellen Lernens (ML) kann dazu beitragen, diese Belastung zu verringern, indem sie Instruments ermöglicht, die es Ärzten ermöglichen, diese Bilder genauer und effizienter zu interpretieren. Die Entwicklung und Implementierung solcher ML-Instruments wird jedoch häufig durch die Verfügbarkeit hochwertiger Daten, ML-Experience und Rechenressourcen eingeschränkt.
Eine Möglichkeit, den Einsatz von ML für die medizinische Bildgebung zu katalysieren, sind domänenspezifische Modelle, die Deep Studying (DL) nutzen, um die Informationen in medizinischen Bildern als komprimierte numerische Vektoren (sogenannte Einbettungen) zu erfassen. Diese Einbettungen stellen eine Artwork vorab erlerntes Verständnis der wichtigen Merkmale in einem Bild dar. Das Erkennen von Mustern in den Einbettungen reduziert im Vergleich zu Arbeiten mit hochdimensionalen Daten, wie zum Beispiel Bilder, direkt. Tatsächlich können diese Einbettungen verwendet werden, um eine Vielzahl nachgelagerter Aufgaben innerhalb der Fachdomäne auszuführen (siehe animierte Grafik unten). Dieser Rahmen, bei dem vorerlerntes Verständnis genutzt wird, um verwandte Aufgaben zu lösen, ähnelt dem eines erfahrenen Gitarristen, der schnell ein neues Lied nach Gehör lernt. Da der Gitarrist bereits eine Grundlage an Können und Verständnis aufgebaut hat, kann er sich schnell die Muster und den Groove eines neuen Songs aneignen.
Um diese Artwork von Einbettungsmodell verfügbar zu machen und die Weiterentwicklung von ML-Instruments in der medizinischen Bildgebung voranzutreiben, freuen wir uns, zwei domänenspezifische Instruments für Forschungszwecke freizugeben: Derm-Stiftung Und Path Basis. Dies folgt auf die starke Resonanz, die wir bereits von Forschern erhalten haben, die das verwenden CXR-Stiftung Einbettungstool für Bruströntgenaufnahmen und stellt einen Teil unseres wachsenden Forschungsangebots für mehrere medizinisch spezialisierte Modalitäten dar. Diese Einbettungswerkzeuge nehmen ein Bild als Eingabe und erzeugen einen numerischen Vektor (die Einbettung), der auf die Bereiche Dermatologie bzw. digitale Pathologiebilder spezialisiert ist. Durch die Ausführung eines Datensatzes mit Thorax-Röntgenbildern, Dermatologie- oder Pathologiebildern über das entsprechende Einbettungstool können Forscher Einbettungen für ihre eigenen Bilder erhalten und diese Einbettungen nutzen, um schnell neue Modelle für ihre Anwendungen zu entwickeln.
Path Basis
In „Domänenspezifische Optimierung und vielfältige Bewertung selbstüberwachter Modelle für die Histopathologie„Wir haben gezeigt, dass selbstüberwachte Lernmodelle (SSL) für Pathologiebilder herkömmliche Vortrainingsansätze übertreffen und ein effizientes Coaching von Klassifikatoren für nachgelagerte Aufgaben ermöglichen. Diese Bemühungen konzentrierten sich auf Hämatoxylin und Eosin (H&E)-gefärbte Objektträger, die wichtigste Gewebefärbung in der diagnostischen Pathologie, die es Pathologen ermöglicht, zelluläre Merkmale unter einem Mikroskop sichtbar zu machen. Die Leistung linearer Klassifikatoren, die mithilfe der Ausgabe der SSL-Modelle trainiert wurden, entsprach der Leistung früherer DL-Modelle, die auf um Größenordnungen besser gekennzeichneten Daten trainiert wurden.
Aufgrund erheblicher Unterschiede zwischen digitalen Pathologiebildern und „natürlichen Bild“-Fotos umfasste diese Arbeit mehrere pathologiespezifische Optimierungen während des Modelltrainings. Ein Schlüsselelement ist das ganze Folienbilder (WSIs) in der Pathologie können einen Durchmesser von 100.000 Pixeln haben (tausende Male größer als typische Smartphone-Fotos) und werden von Experten bei mehreren Vergrößerungen (Zoomstufen) analysiert. Daher werden die WSIs typischerweise in kleinere Kacheln oder Patches für Pc Imaginative and prescient- und DL-Anwendungen unterteilt. Die resultierenden Bilder weisen eine hohe Informationsdichte mit über das Bild verteilten Zellen oder Gewebestrukturen auf, anstatt unterschiedliche semantische Objekte oder Vorder- und Hintergrundvariationen aufzuweisen, was einzigartige Herausforderungen für robustes SSL und Merkmalsextraktion mit sich bringt. Darüber hinaus sind körperliche (z. B. Schneiden) und chemisch (z. B. Festsetzung Und Fleckenbildung) Prozesse, die zur Vorbereitung der Proben verwendet werden, können das Erscheinungsbild des Bildes dramatisch beeinflussen.
Unter Berücksichtigung dieser wichtigen Aspekte umfassten pathologiespezifische SSL-Optimierungen die Unterstützung des Modells beim Lernen fleckenunabhängige MerkmaleVerallgemeinerung des Modells auf Patches aus mehreren Vergrößerungen, verstärkend die Daten zur Nachahmung des Scannens und der Bildnachbearbeitung sowie benutzerdefinierter Datenausgleich, um die Eingabeheterogenität für das SSL-Coaching zu verbessern. Diese Ansätze wurden anhand einer breiten Palette von Benchmark-Aufgaben umfassend evaluiert, an denen 17 verschiedene Gewebetypen in 12 verschiedenen Aufgaben beteiligt waren.
Verwendung des Imaginative and prescient-Transformators (ViT-S/16)-Architektur wurde Path Basis aus dem oben beschriebenen Optimierungs- und Bewertungsprozess als Modell mit der besten Leistung ausgewählt (und in der Abbildung unten dargestellt). Dieses Modell bietet somit ein wichtiges Gleichgewicht zwischen Leistung und Modellgröße, um eine wertvolle und skalierbare Verwendung bei der Generierung von Einbettungen über die vielen einzelnen Bildfelder großer pathologischer WSIs zu ermöglichen.
 |
| SSL-Coaching mit pathologiespezifischen Optimierungen für Path Basis. |
Der Wert domänenspezifischer Bilddarstellungen ist auch in der folgenden Abbildung ersichtlich, die die lineare Sondierungsleistungsverbesserung von Path Basis zeigt (gemessen durch). AUROC) im Vergleich zum herkömmlichen Vortraining auf natürlichen Bildern (ImageNet-21k). Dazu gehört die Auswertung von Aufgaben wie z.B Erkennung von metastasiertem Brustkrebs in Lymphknoten, Einstufung von ProstatakrebsUnd Einstufung von Brustkrebsunter anderen.
 |
| Path Basis-Einbettungen übertreffen herkömmliche ImageNet-Einbettungen deutlich, wenn sie durch lineare Sondierung über mehrere Bewertungsaufgaben in der Histopathologie hinweg bewertet werden. |
Derm-Stiftung
Derm-Stiftung ist ein Einbettungstool, das aus unserer Forschung zur Anwendung von DL abgeleitet wurde Bilder von dermatologischen Erkrankungen interpretieren und enthält unsere neuesten Arbeiten, die hinzufügen Verbesserungen zur besseren Verallgemeinerung auf neue Datensätze. Aufgrund seiner dermatologiespezifischen Vorschulung verfügt es über ein latentes Verständnis der in Bildern von Hauterkrankungen vorhandenen Merkmale und kann zur schnellen Entwicklung von Modellen zur Klassifizierung von Hauterkrankungen verwendet werden. Das der API zugrunde liegende Modell ist a BiT ResNet-101×3 in zwei Stufen trainiert. Die erste Vortrainingsphase nutzt kontrastives Lernen, ähnlich wie ConVIRTum eine große Anzahl von Bild-Textual content-Paaren zu trainieren aus dem Web. Im zweiten Schritt wird die Bildkomponente dieses vorab trainierten Modells dann für die Zustandsklassifizierung anhand klinischer Datensätze, beispielsweise von teledermatologischen Diensten, verfeinert.
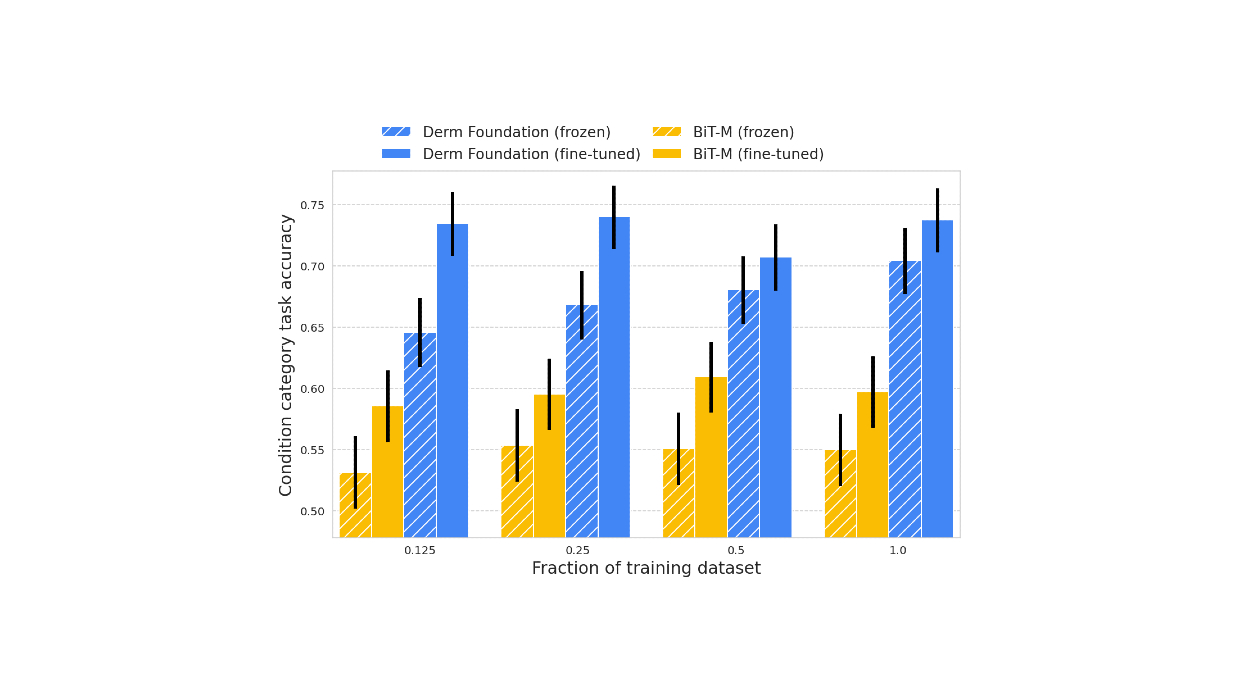
Im Gegensatz zu histopathologischen Bildern ähneln dermatologische Bilder eher den realen Bildern, die zum Trainieren vieler heutiger Pc-Imaginative and prescient-Modelle verwendet werden. Für spezielle dermatologische Aufgaben ist jedoch möglicherweise immer noch ein großer Datensatz erforderlich, um ein qualitativ hochwertiges Modell zu erstellen. Mit Derm Basis können Forscher ihren eigenen kleineren Datensatz verwenden, um domänenspezifische Einbettungen abzurufen, und diese verwenden, um kleinere Modelle (z. B. lineare Klassifikatoren oder andere kleine nichtlineare Modelle) zu erstellen, die es ihnen ermöglichen, ihre Forschungs- oder Produktideen zu validieren. Um diesen Ansatz zu bewerten, haben wir Modelle anhand von Teledermatologiedaten für eine nachgelagerte Aufgabe trainiert. Das Modelltraining umfasste unterschiedliche Datensatzgrößen (12,5 %, 25 %, 50 %, 100 %), um einbettungsbasierte lineare Klassifikatoren mit der Feinabstimmung zu vergleichen.
Als Modellierungsvarianten wurden berücksichtigt:
- Ein linearer Klassifikator für eingefrorene Einbettungen von BiT-M (ein standardmäßiges vorab trainiertes Bildmodell)
- Feinabgestimmte Model von BiT-M mit einer besonders dichten Schicht für die nachgelagerte Aufgabe
- Ein linearer Klassifikator für eingefrorene Einbettungen aus der Derm Basis API
- Feinabgestimmte Model des Modells, das der Derm Basis API zugrunde liegt, mit einer zusätzlichen Ebene für die nachgelagerte Aufgabe
Wir haben festgestellt, dass Modelle, die auf den Einbettungen der Derm Basis für dermatologische Aufgaben basieren, eine deutlich höhere Qualität erzielen als Modelle, die ausschließlich auf Einbettungen basieren oder mithilfe von BiT-M verfeinert werden. Es wurde festgestellt, dass dieser Vorteil bei kleineren Trainingsdatensatzgrößen am ausgeprägtesten ist.
Allerdings gibt es bei dieser Analyse Einschränkungen. Wir untersuchen immer noch, wie intestine sich diese Einbettungen über Aufgabentypen, Patientenpopulationen und Bildeinstellungen hinweg verallgemeinern lassen. Nachgeschaltete Modelle, die mit Derm Basis erstellt wurden, erfordern noch eine sorgfältige Bewertung, um ihre erwartete Leistung in der beabsichtigten Umgebung zu verstehen.
Entry Path und Derm Basis
Wir gehen davon aus, dass die Einbettungstools der Derm Basis und Path Basis eine Reihe von Anwendungsfällen ermöglichen werden, darunter die effiziente Entwicklung von Modellen für Diagnoseaufgaben, Verbesserungen der Qualitätssicherung und voranalytischen Arbeitsabläufe, Bildindizierung und -kuration sowie die Entdeckung und Validierung von Biomarkern. Wir stellen beide Instruments der Forschungsgemeinschaft zur Verfügung, damit diese den Nutzen der Einbettungen für ihre eigenen Dermatologie- und Pathologiedaten untersuchen kann.
Um Zugriff zu erhalten, melden Sie sich bitte über die folgenden Google-Formulare für die Nutzungsbedingungen der einzelnen Instruments an.
Nachdem Sie Zugriff auf jedes Instrument erhalten haben, können Sie die API verwenden, um Einbettungen aus Dermatologiebildern oder digitalen Pathologiebildern abzurufen, die in Google Cloud gespeichert sind. Zugelassene Benutzer, die nur neugierig sind, das Modell und die Einbettungen in Aktion zu sehen, können die bereitgestellten Colab-Beispielnotizbücher verwenden, um Modelle mithilfe öffentlicher Daten zur Klassifizierung zu trainieren Sechs häufige Hauterkrankungen oder die Identifizierung von Tumoren Histopathologische Pflaster. Wir freuen uns darauf, die Bandbreite der Anwendungsfälle zu sehen, die diese Instruments ermöglichen.
Danksagungen
Wir möchten den vielen Mitarbeitern danken, die dazu beigetragen haben, diese Arbeit möglich zu machen, darunter Yun Liu, Can Kirmizi, Fereshteh Mahvar, Bram Sterling, Arman Tajback, Kenneth Philbrik, Arnav Agharwal, Aurora Cheung, Andrew Sellergren, Boris Babenko, Basil Mustafa und Jan Freyberg , Terry Spitz, Yuan Liu, Pinal Bavishi, Ayush Jain, Amit Talreja, Rajeev Rikhye, Abbi Ward, Jeremy Lai, Faruk Ahmed, Supriya Vijay, Tiam Jaroensri, Jessica Lavatory, Saurabh Vyawahare, Saloni Agarwal, Ellery Wulczyn, Jonathan Krause, Fayaz Jamil, Tom Small, Annisah Um’rani, Lauren Winer, Sami Lachgar, Yossi Matias, Greg Corrado und Dale Webster.